Page History
Krótki opis usługi
Usługa InSilicoLab ma na celu wsparcie uruchamiania złożonych eksperymentów obliczeniowych chemii kwantowej na infrastrukturze PL-Grid. Pozwala na przygotowanie danych wejściowych (w tym serii plików o wspólnej strukturze) dla wspieranych programów kwantowochemicznych, wysłanie przygotowanych zadań na grid i ich uruchomienie, a następnie zebranie plików wynikowych i ich wstępną analizę. Przygotowywanie danych wejściowych do usługi InSilicoLab for Chemistry może zostać dodatkowo ułatwione poprzez użycie narzędzia Quantum Chemistry Advisor.
Dzięki zapisywaniu danych w katalogach sieciowych, możliwe jest przekazywanie danych z jednego eksperymentu obliczeniowego do drugiego oraz dostęp do nich niezależny od komputera, z którego zlecono zadania. Możliwość zapamiętania eksperymentu pozwala na łatwe odtworzenie cyklu obliczeniowego.
...
Obecnie dostępne są następujące eksperymenty InSilicoLab:
Obliczenia kwantowochemiczne
Eksperyment ten pozwala przygotowanie plików danych wejściowych dla kilku programów obliczeniowych chemii kwantowej (Gaussian, Gamess, Turbomole, Niedoida), specyfikację żądań zasobów a następnie uruchomienie obliczeń na infrastrukturze gridowej.
Po zakończeniu obliczeń użytkownik ma możliwość pobrania plików wynikowych, przeprowadzenia wstępnej analizy (w tym uzyskania zestawienia obliczonych parametrów w formacie XML) oraz zachowania danych i wyników w przestrzeni storage'owej w celu dalszego wykorzystania w kolejnych eksperymentach.
Trajectory Sculptor
Trajectory Sculptor jest narzędziem ułatwiającym manipulacje danymi podczas sekwencyjnego modelowania kwantowochemicznego złożonych układów, w szczególności cząsteczek w roztworach.
...
Niezbędnymi danymi wejściowymi jest trajektoria dynamiki molekularnej i informacja o warunkach brzegowych. Użytkownik ma do dyspozycji wiele możliwości specyfikacji otoczki solwatacyjnej wycinanej wraz z cząsteczką rozpuszczoną dzięki różnym sposobom określania odległości rozpuszczalnik-cząsteczka rozpuszczona oraz wybierania wycinanych cząsteczek rozpuszczalnika (do zadanej odległości lub zadana liczba cząsteczek). Na każdym etapie pracy możliwa jest wizualizacja wyników. Efektem działania narzędzia są geometrie układów wyciętych z poszczególnych ramek zapisane w osobnych plikach lub jako trajektoria w pojedynczym pliku. Możliwe jest też przekazanie ich do innego eksperymentu InSilicoLab w celu uruchomienia obliczeń na infrastrukturze PL-Grid.
Eksperyment Cubegen
Służy do generowania tzw. plików cube programu Gaussian. Niezbędne jest dostarczenie (np. z przestrzeni storage'owej użytkownika) wygenerowanego wcześniej (np. w ogólnym eksperymencie InSilicoLab) pliku checkpoint z przechowanymi danymi obliczeń.
...
TeraChem
Eksperyment ten ułatwia użycie programu TeraChem służącego do wykonywania obliczeń kwantowochemicznych na kartach graficznych ogólnego przeznaczenia (GP GPU computing). Pozwala to na znaczne, nawet kilkunastokrotne przyspieszenie niektórych obliczeń (w zależności od rozmiaru układu i stosowanej metody).
| Anchor | ||||
|---|---|---|---|---|
|
Aby skorzystać z usługi InSilicoLab for Chemistry należy aktywować ją w katalogu aplikacji i usług.
Dodatkowo, do wykonywania obliczeń kwantowochemicznych, wymagane jest aktywowanie usługi "Globalny dostęp gLite
...
Aby skorzystać z usługi InSilicoLab for Chemistry należy aktywować ją w Portalu PL-Grid. Jest ona dostępna w katalogu usług w dziale "Platforma dziedzinowa: chemia".
Dodatkowo, do wykonywania obliczeń kwantowochemicznych, wymagane jest aktywowanie usługi "Globalny dostęp gLite" dostępnej w katalogu usług w dziale "Usługi globalne". Aktywowanie tej usługi może się odbyć wyłącznie po zarejestrowaniu conajmniej co najmniej jednego certyfikatu osobistego w w Portalu PL-Grid - informację na temat temat rejestracji oraz oraz możliwości uzyskania certyfikatu można znaleźć w rozdziale "Aplikowanie, rejestracja i użycie certyfikatu" niniejszego Pierwsze kroki w Portalu Użytkownika" niniejszego podręcznika.
Uwaga: Zaraz po aktywowaniu usługi "Globalny dostęp gLite" informacje na temat konta użytkownika muszą zostać przekazane do infrastruktury - ten proces może trwać maksymalnie do 6 godzin. W tym czasie może nie być dostępna pełna funkcjonalność eksperymentów kwantowochemicznych (uruchamianie eksperymentów oraz pobieranie danych). Po spropagowaniu wszystkich danych pełna funkcjonalność systemu jest dostępna.
Informacje dotyczące usług i ich aktywowania można znaleźć w rozdziale "Usługi" niniejszego podręcznika.
Pierwsze kroki
Należy połączyć się z serwerem usługi
http://insilicolab.chemia.plgrid.pl/
Po wejściu do usługi, otwiera się ekran. Jeśli nie byliśmy wcześniej zalogowani do usługi, dostępny jest dla nas jedynie ekran anonimowego użytkownika. Logowanie do własnego konta następuje po przyciśnięciu przycisku Log in... i jest możliwe jest na dwa sposoby:
- Logowanie poprzez certyfikat zarejestrowany w przeglądarce użytkownika (nasze dane zostaną sczytane z certyfikatu)
- Logowanie poprzez OpenID dla infrastruktury PL-Grid. W tym wypadku zostaniemy przekierowani na stronę OpenID dla PL-Grid i poproszeni o wpisanie danych dostępowych do infrastruktury - nazwy użytkownika i hasła (tych samych które podajemy w Portalu PL-Grid) lub sczytany zostanie certyfikat umieszczony w przeglądarce. Jeśli usługa InSilicoLab for Chemistry nie była aktywna wcześniej zostanie ona aktywowana automatycznie po zaakceptowaniu odpowiedniego komunikatu. Po zalogowaniu zostaniemy automatycznie przekierowani z powrotem na stronę usługi.
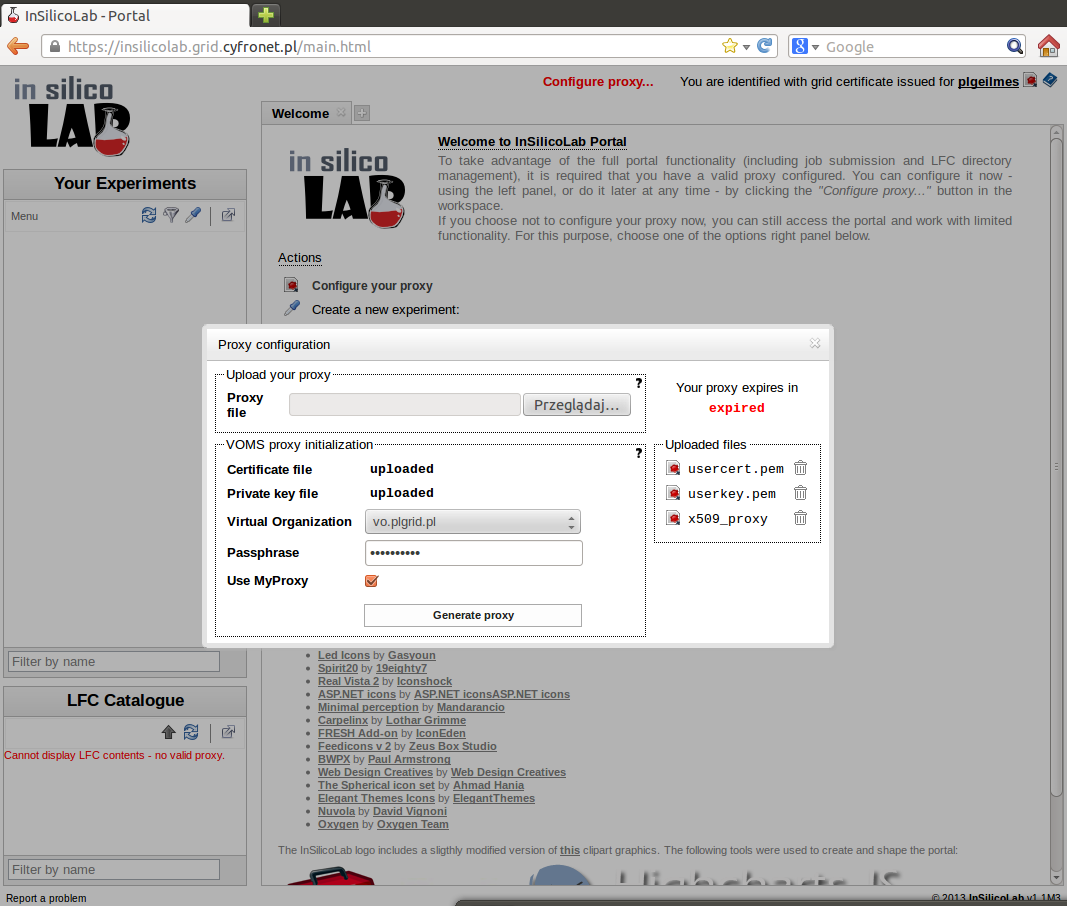
W celu przeprowadzenia obliczeń niezbędne będzie skonfigurowanie odpowiedniego certyfikatu proxy przez kliknięcie na link Configure your proxy:
Certyfikat proxy konieczny jest do przeprowadzania obliczeń z wykorzystaniem infrastruktury PL-Grid a także przeglądania danych zgromadzonych w zasobach tej infrastruktury. Nie jest natomiast konieczny do przeglądania konta użytkownika i zapisanych przez niego analiz a także do wstępnego przygotowania danych przy pomocy narzędzia Trajectory Sculptor (patrz rozdział "Zaawansowane użycie").
Istnieją dwie możliwości konfiguracji proxy w portalu:
- Załadowanie istniejącego proxy - wygenerowanego uprzednio na infrastrukturze. Instrukcja jak wygenerować takie proxy dostępna jest po kliknięciu w pytajnik w polu Upload your proxy. Jest to opcja bezpieczniejsza dla użytkownika, ale wymaga każdorazowego ładowania proxy po upływie jego ważności (12 godzin).
- Generacja proxy z poziomu portalu. Aby wygenerować proxy w portalu, należy załadować do niego pliki certyfikatu i klucza w formacie PEM (najczęściej są to usercert.pem i userkey.pem) lub PKCS12 (np. usercert.p12). Jest to czynność jednorazowa, i nie będzie potrzeby ponawiania jej w przyszłości jeśli certyfikat użytkownika nie ulegnie zmianie. Następnie należy wybrać wirtualną organizację - dla infrastruktury PL-Grid jest to vo.plgrid.pl, podać hasło do załadowanego klucza (UWAGA: hasło nie jest przechowywane nigdzie w systemie) oraz kliknąć przycisk Generate proxy. Zaznaczenie opcji Use MyProxy da nam możliwość automatycznego przedłużania certyfikatu proxy dla uruchomionych zadań do 168. Dodatkowe instrukcje (m. in. instrukcje postępowania w przypadku kiedy certyfikat użytkownika jest w formacie innym niż PEM) dostępne są po kliknięciu w pytajnik w polu VOMS proxy initialization.
Wykonywanie obliczeń z użyciem pakietu TeraChem wymaga także aktywowania dodatkowych usług:
- Dostęp do klastra ZEUS - Cyfronet
- Dostęp do GPGPU - Cyfronet
- PLG-Data: mini-usługa do zarządzania plikami na klastrze
- Rimrock
Zwracamy również uwagę że, w związku z przejściem na nowy system scratch na maszynie Zeus, dla osób które używały starego systemu scratch, konieczne jest przełączenie na nowy. Sprawdzenia, który system jest używany można dokonać wyświetlając zmienną $SCRATCH (echo $SCRATCH) - powinna ona wskazywać /mnt/lustre/scratch2/people/<nazwa_użytkownika>. Przełączenia na nowy system scratch można dokonać w następujący sposób:
- Należy zalogować się przy użyciu ssh na maszynę zeus.cyfronet.pl (nazwa użytkownika i konta jest tożsama z użytkownikiem i kontem w portalu http://portal.plgrid.pl)
- Usunąć plik ~/.zeusoldscratch (
rm ~/.zeusoldscratch) - Zalogować się ponownie
- Sprawdzić czy zmienna $SCRATCH wskazuje właściwą lokalizację (
echo $SCRATCH) -/mnt/lustre/scratch2/people/<nazwa_użytkownika>
Informacje dotyczące usług i ich aktywowania można znaleźć w podręczniku użytkownika.
Pierwsze kroki
Należy połączyć się z serwerem usługi
https://insilicolab.chemia.plgrid.pl/
Po wejściu do usługi, otwiera się ekran. Jeśli nie byliśmy wcześniej zalogowani do usługi, dostępny jest dla nas jedynie ekran anonimowego użytkownika. Logowanie do własnego konta następuje po przyciśnięciu przycisku Log in... i jest możliwe jest na dwa sposoby:
- Logowanie poprzez certyfikat zarejestrowany w przeglądarce użytkownika (nasze dane zostaną sczytane z certyfikatu)
- Logowanie poprzez OpenID dla infrastruktury PL-Grid. W tym wypadku zostaniemy przekierowani na stronę OpenID dla PL-Grid i poproszeni o wpisanie danych dostępowych do infrastruktury - nazwy użytkownika i hasła (tych samych które podajemy w Portalu PL-Grid) lub sczytany zostanie certyfikat umieszczony w przeglądarce. Jeśli usługa InSilicoLab for Chemistry nie była aktywna wcześniej zostanie ona aktywowana automatycznie po zaakceptowaniu odpowiedniego komunikatu. Po zalogowaniu zostaniemy automatycznie przekierowani z powrotem na stronę usługi.
W celu przeprowadzenia obliczeń niezbędne będzie skonfigurowanie odpowiedniego certyfikatu proxy przez kliknięcie na link Configure your proxy:
Certyfikat proxy konieczny jest do przeprowadzania obliczeń z wykorzystaniem infrastruktury PL-Grid a także przeglądania danych zgromadzonych w zasobach tej infrastruktury. Nie jest natomiast konieczny do przeglądania konta użytkownika i zapisanych przez niego analiz a także do wstępnego przygotowania danych przy pomocy narzędzia Trajectory Sculptor (patrz rozdział "Zaawansowane użycie").
Istnieją dwie możliwości konfiguracji proxy w portalu:
- Załadowanie istniejącego proxy - wygenerowanego uprzednio na infrastrukturze. Instrukcja jak wygenerować takie proxy dostępna jest po kliknięciu w pytajnik w polu Upload your proxy. Jest to opcja bezpieczniejsza dla użytkownika, ale wymaga każdorazowego ładowania proxy po upływie jego ważności (12 godzin).
- Generacja proxy z poziomu portalu. Aby wygenerować proxy w portalu, należy załadować do niego pliki certyfikatu i klucza w formacie PEM (najczęściej są to usercert.pem i userkey.pem) lub PKCS12 (np. usercert.p12). Jest to czynność jednorazowa, i nie będzie potrzeby ponawiania jej w przyszłości jeśli certyfikat użytkownika nie ulegnie zmianie. Następnie należy wybrać wirtualną organizację - dla infrastruktury PL-Grid jest to vo.plgrid.pl, podać hasło do załadowanego klucza (UWAGA: hasło nie jest przechowywane nigdzie w systemie) oraz kliknąć przycisk Generate proxy. Zaznaczenie opcji Use MyProxy da nam możliwość automatycznego przedłużania certyfikatu proxy dla uruchomionych zadań do 168. Dodatkowe instrukcje (m. in. instrukcje postępowania w przypadku kiedy certyfikat użytkownika jest w formacie innym niż PEM) dostępne są po kliknięciu w pytajnik w polu VOMS proxy initialization.
W W obu przypadkach, jeśli proxy zostało skonfigurowane poprawnie, zostanie wyświetlona na zielono ilość pozostałego czasu ważności proxy w portalu (w polu Your proxy expires in).
...
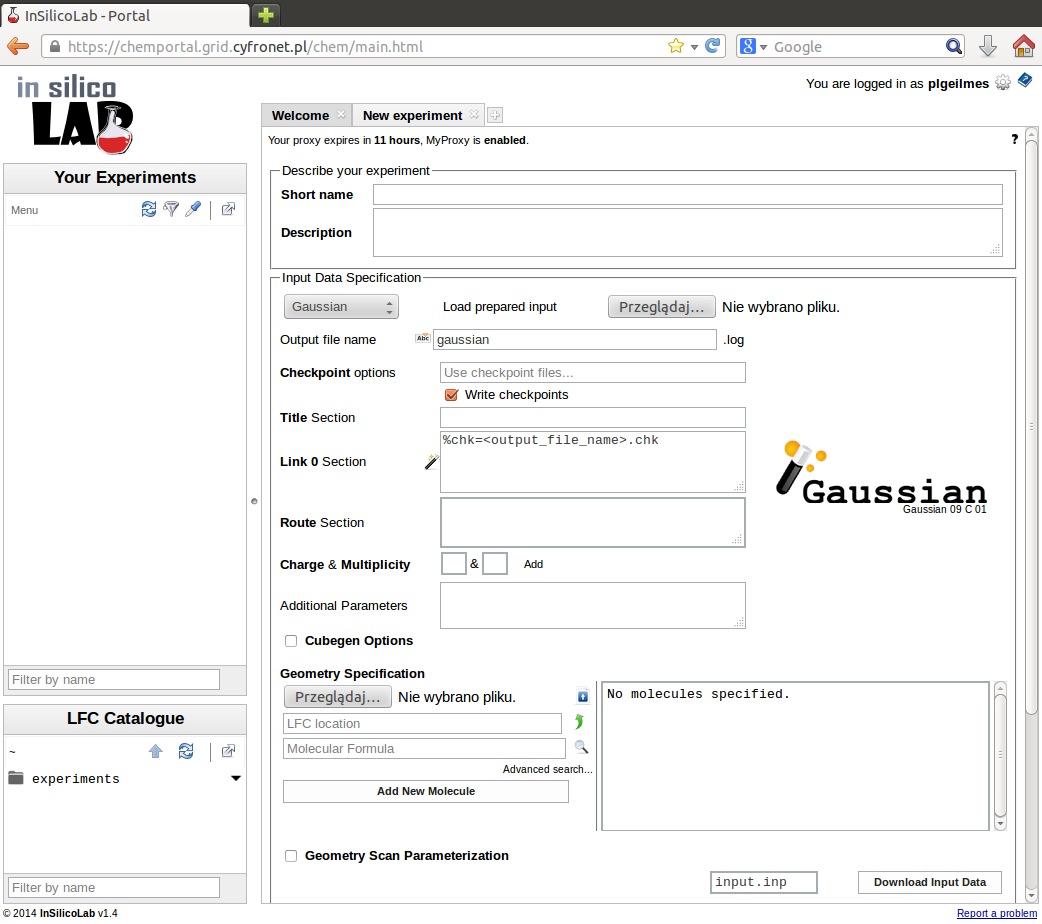
W przeglądarce otworzy się karta nowego eksperymentu obliczeniowego wykorzystującego programy programu chemii kwantowej.
Uwaga: przy wyborze odpowiedniego programu chemii kwantowej, a także przy uzupełnianiu jego parametrów, można skorzystać z usługi Quantum Chemistry Advisor, agregującej dane o tych programach i wspomagającej wybór odpowiedniego do danych obliczeń.
Identyfikację Identyfikację eksperymentu wprowadzamy podając jego krótką nazwę oraz (opcjonalnie) dłuższy opis. W przypadku niepodania krótkiej nazwy, wykorzystane zostanie pole Title formatki aplikacji kwantowochemicznej. Jeśli oba pola zostaną puste, jako krótka nazwa użyty zostanie tekst "(no title)".
...
Parametry i dane zakończonych zadań mogą zostać zachowane w spisie eksperymentów użytkownika, w celu późniejszego wykorzystania lub powtórzenia.
Zaawansowane użycie
Po połączeniu się z serwerem usługi
httphttps://insilicolab.chemia.plgrid.pl
przejściu do portalu InSilicoLab i skonfigurowaniu proxy (patrz Pierwsze kroki) z menu Create a new experiment wybieramy pozycję Trajectory Sculptor. W przeglądarce otworzy się karta eksperymentu pozwalającego przygotować dane do obliczeń kwantowochemicznych poprzez wycięcie wybranych fragmentów układu z ramek trajektorii dynamiki molekularnej.
...
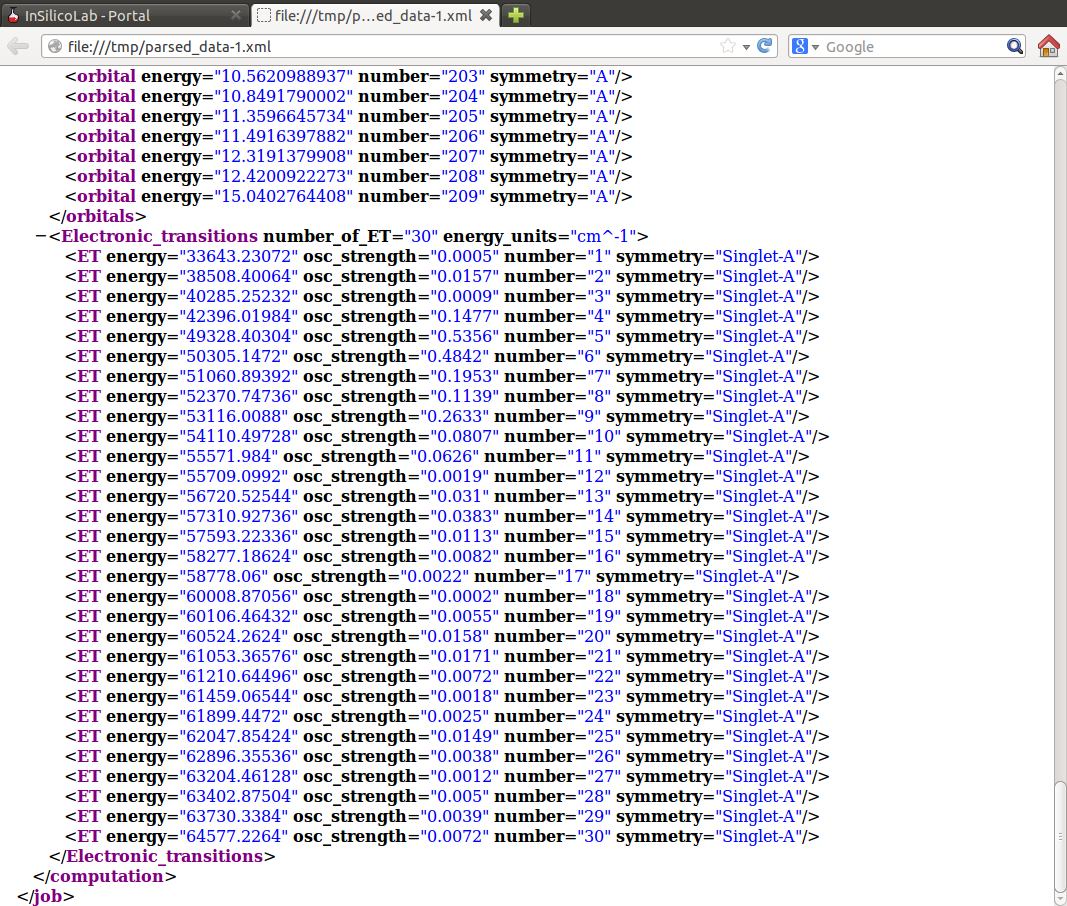
W szczególności dla obliczeń widm elektronowych plik ten zawiera dane o energii przejść oraz ich sile oscylatora:
Użycie programu niedoida w eksperymencie kwantowochemicznym
Program niedoida rozwijany jest na Wydziale Chemii UJ we współpracy z Akademickim Centrum Komputerowym ACK Cyfronet AGH. Implementuje standardowe metody chemii kwantowej (HF, MP2, metody DFT i TD DFT). Unikatowymi cechami programu jest implementacja Laplace-Transform MP2 oraz dressed-TD DFT.
...
units: length: angstrom energy: hartreemoller_plesset: order: 2 type: laplace_ao quadrature: fitted_simple
Określiliśmy jednostki odległości i energii oraz zażądaliśmy obliczeń post-HF metodą MP2 z wykorzystaniem transformacji Laplace'a.
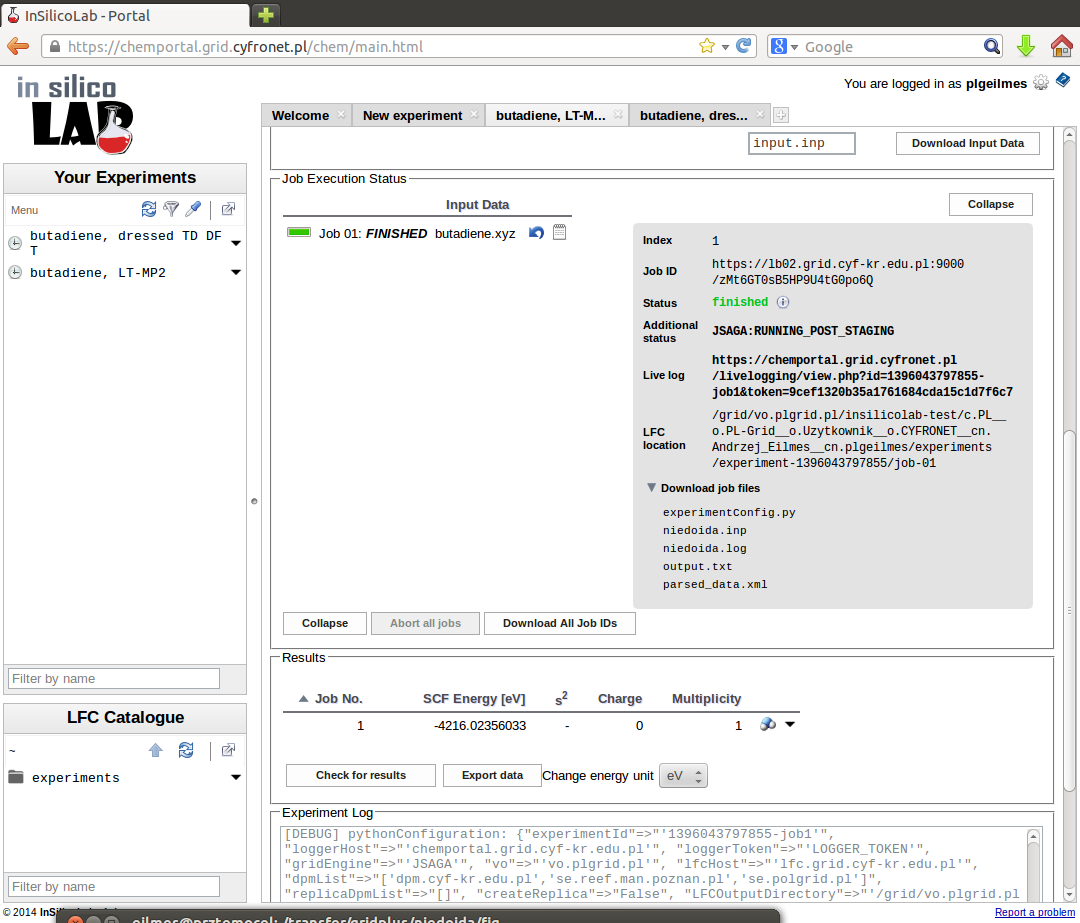
Obliczenia uruchamiamy kliknięciem w przycisk Run. Po ich zakończeniu możemy ściągnąć plik outputowy (niedoida.log) oraz plik XML ze sparsowanymi wynikami.
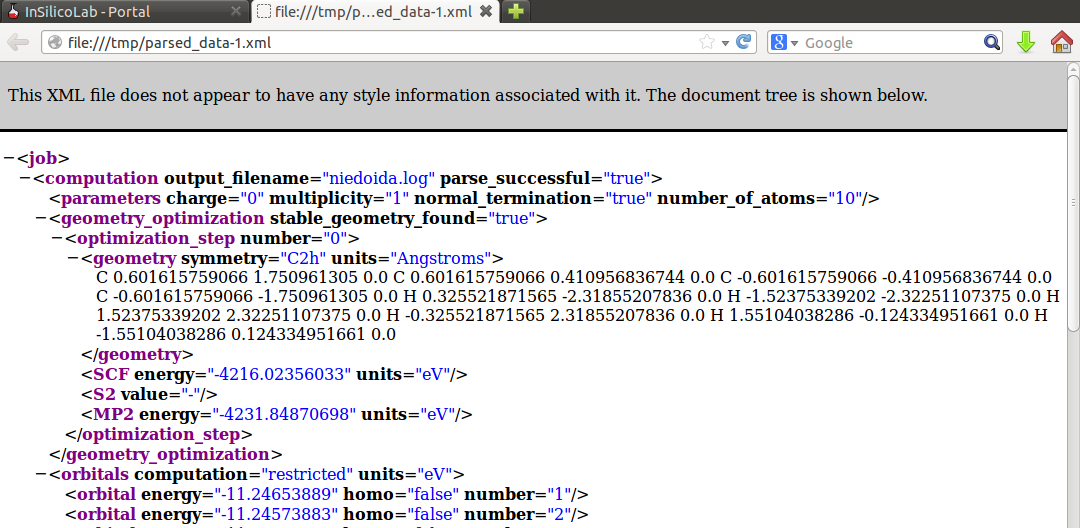
Zwróćmy uwagę, że w polu Results podawana jest energia na poziomie HF SCF, zatem aby odczytać interesującą nas energię w metodzie MP2 należy obejrzeć output programu, lub (wygodniej) odczytać wartość z pliku parsed_data.xml ("MP2 energy"):
: fitted_simple
Określiliśmy jednostki odległości i energii oraz zażądaliśmy obliczeń post-HF metodą MP2 z wykorzystaniem transformacji Laplace'a.
Obliczenia uruchamiamy kliknięciem w przycisk Run. Po ich zakończeniu możemy ściągnąć plik outputowy (niedoida.log) oraz plik XML ze sparsowanymi wynikami.
Zwróćmy uwagę, że w polu Results podawana jest energia na poziomie HF SCF, zatem aby odczytać interesującą nas energię w metodzie MP2 należy obejrzeć output programu, lub (wygodniej) odczytać wartość z pliku parsed_data.xml ("MP2 energy"):
Obliczenia kwantowochemiczne na GPU (program TeraChem)
Dostępny na klastrze Zeus program TeraChem pozwala na przyspieszenie części typowych obliczeń kwantowochemicznych poprzez wykonywanie ich na kartach graficznych NVidia.
Uwaga: aby móc skorzystać z obliczeń TeraChemem, należy aktywować dodatkowe usługi - patrz rozdział Aktywowanie usługi.
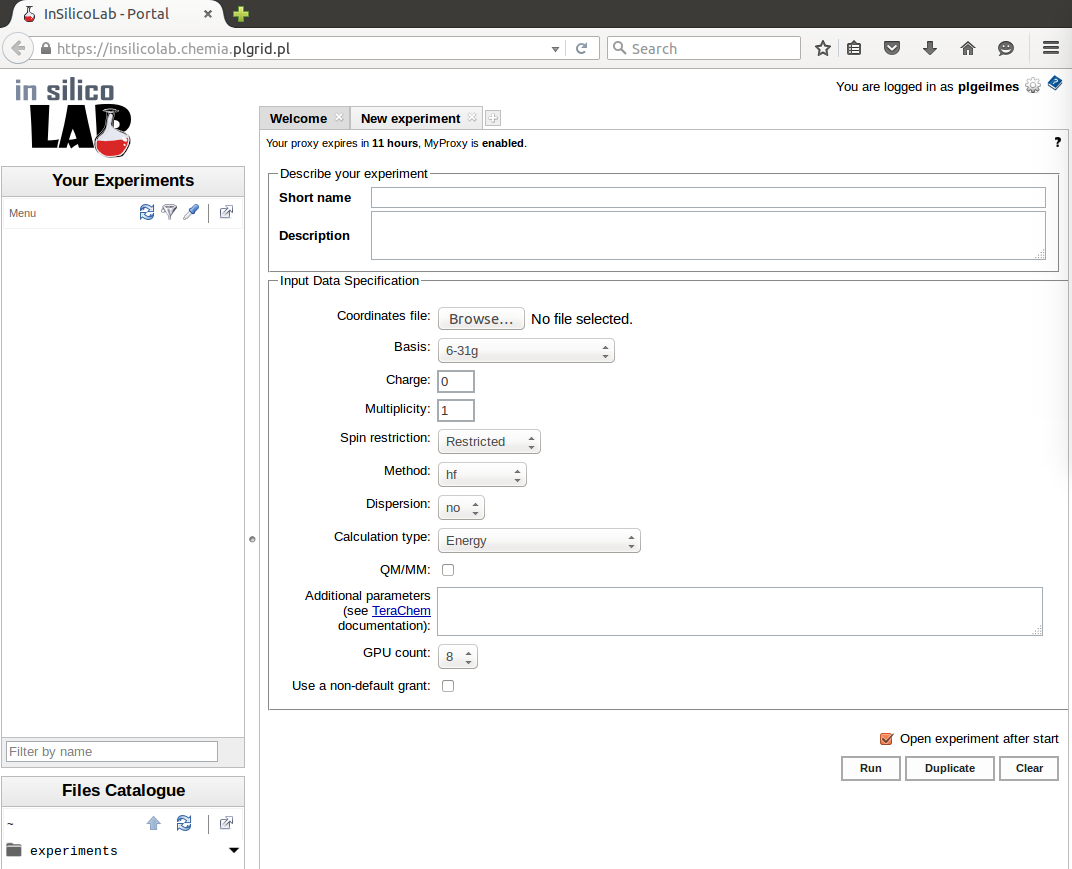
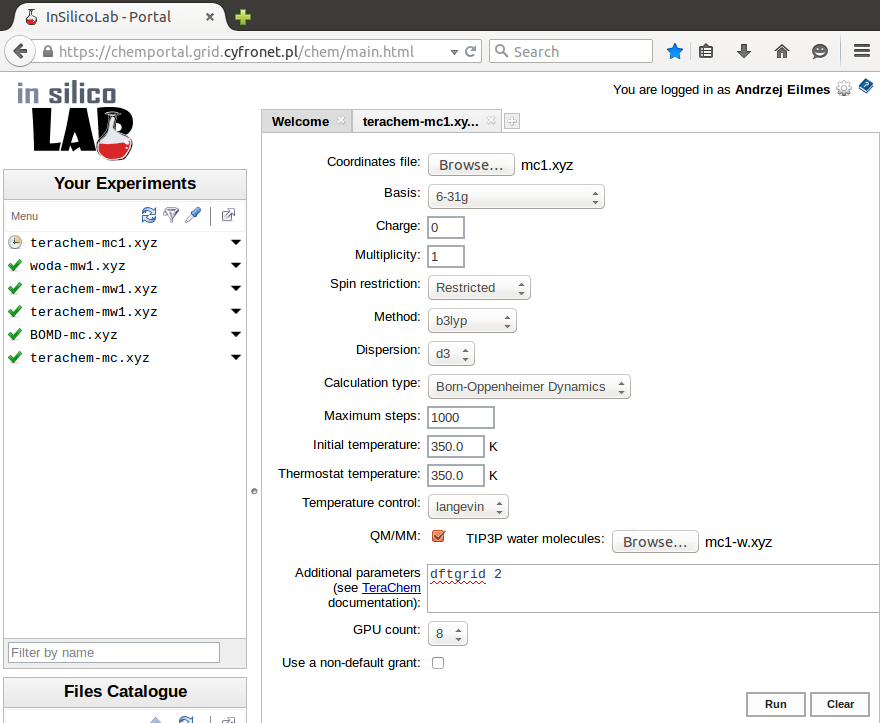
Szablon odpowiedniego eksperymentu wywołuje się klikając nazwę TeraChem w głównym menu.
Parametry możliwe do ustawienia w eksperymencie to:
- Coordinates file - pozwala na załadowanie pliku w formacie
xyzzawierającego geometrię układu - Basis - baza funkcyjna żądana w obliczeniach
- Charge - całkowity ładunek układu
- Multiplicity - multipletowość układu
- Spin restriction - typ obliczeń restricted lub unrestricted. W przypadku, gdy multipletowość jest większa od 1, wybór typu restricted oznacza uruchomienie obliczeń restricted open-shell.
- Method - metoda kwantowochemiczna. Możliwe jest wykonywanie obliczeń metodą Hartree-Focka lub obliczeń DFT z użyciem różnych funkcjonałów
- Dispersion - pozwala uwzględnić w obliczeniach empiryczną poprawkę dyspersyjną Grimmego (w wersji D2 lub D3)
- Calculation type - rodzaj obliczeń: obliczenia energii (single point), optymalizacja geometrii, poszukiwanie stanu przejściowego, dynamika Borna-Oppenheimera, obliczenie energii i gradientu oraz projekcja funkcji falowej
- QM/MM - zaznaczenie tej opcji pozwala na uwzględnienie wody traktowanej metodą mechaniki molekularnej
- Additional parameters - pole pozwalające na podanie innych słów kluczowych programu
- GPU count - liczba kart graficznych, na których ma zostać uruchomiony program (od 1 do 8)
- Use a non-default grant - standardowo obliczenia wykonywane są w ramach zasobów z domyślnego grantu użytkownika PL Grid. Zaznaczenie tej opcji pozwala na podanie nazwy innego grantu obliczeniowego użytkownika
Jeśli jako Calculation type wybrano Born-Oppenheimer Dynamics, w dodatkowych polach zadaje się:
- Maximum steps - liczbę kroków dynamiki
- Initial temperature - temperaturę określającą początkowy rozkład prędkości
- Thermostat temperature - temperaturę, która ma być utrzymywana w trakcie obliczeń
- Temperature control - sposób utrzymywania zadanej temperatury: termostat Langevina lub skalowanie prędkości
Wybór opcji QM/MM pozwala na wykonanie obliczeń, w których część układu traktowana jest na poziomie wybranej metody kwantowochemicznej a otaczające ją cząsteczki wody opisywane są mechaniką molekularną w parametryzacji TIP3P. W takim przypadku należy użyć listy TIP3P water molecules do załadowania pliku xyz z geometrią cząsteczek wody (część kwantowochemiczna zdefinowana jest przez plik podany w Coordinates file). Cząsteczki wody w pliku zapisywane są kolejno, atomy cząsteczki muszą być podawane w kolejności OHH.
W poniższym przykładzie zażądano wykonania 1000 kroków symulacji dynamiki Borna-Oppenheimera z początkową temperaturą 350 K oraz żądaną temperaturą układu 350 K utrzymywaną przez termostat Langevina. Wybrano kombinowaną metodę QM/MM. Geometria kwantowochemicznej części układu zawarta jest w pliku mc1.xyz, jej ładunek i multipletowość wynoszą 0 i 1. Obliczenia kwantowochemiczne mają zostać wykonane metodą RB3LYP w bazie 6-31G z uwzględnieniem poprawki dyspersyjnej D3. Gęstość gridu używanego w obliczeniach DFT ustawiono na poziomie 2. Kwantowochemiczna część układu otoczona jest cząsteczkami wody TIP3P o początkowej geometrii zadanej w plikumc1-w.xyz. Obliczenia mają być wykonane z użyciem 8 kart GPU w ramach domyślnego grantu użytkownika.
Kliknięcie przycisku Run powoduje przesłanie zadania do wykonania na klastrze Zeus.
Po zakończeniu obliczeń użytkownik ma możliwość pobrania pliku log (output programu TeraChem) oraz pliku tar z zawartością katalogu scr (pozostałe pliki wynikowe tworzone przez program).
...