Page History
| Warning |
|---|
Usługa Biologia: Chipster została wycofana |
Krótki opis usługi
Usługa jest przeznaczona dla biologów oraz bioinformatyków.
Chipster jest implementacją popularnego środowiska zarządzania zadaniami pozwalającego na uproszczone uruchamianie analiz bioinformatycznych na zasobach obliczeniowych PLGrid. W ramach usługi szczególny nacisk położony został na udostępnienie możliwie dużej liczby narzędzi związanych z analizą danych pochodzących z eksperymentów opartych o metody wysokoprzepustowego sekwencjonowania. Wykonywanie analiz z użyciem Chipster oparte jest o wygodny interfejs dostępny w formie aplikacji Java, pozwalający na intuicyjne zarządzanie danymi, narzędziami oraz wynikami. Wbudowane moduły wizualizacji pozwalają na przejrzystą i efektywną analizę wyników.
Aktywowanie usługi
Aby skorzystać z usługi Chipster, należy mieć aktywne konto w Infrastrukturze PLGrid oraz aktywną afiliację.
...
Przyznanie usługi następuje automatycznie. Gdy usługa zostanie przyznana, pojawi się na liście usług w portalu PLGrid ze statusem „active” Status usługi będzie również widoczny w KAiU jako "Status użytkownika: Usługa aktywna — usługa dostępna dla użytkownika".
Pierwsze kroki
Uruchomienie usługi
Usługa Chipster składa się z aplikacji klienckiej Chipster oraz oprogramowania zainstalowanego na serwerach obliczeniowych PLGrid. Aplikacja kliencka służy do uruchamiania oraz zarządzania zadaniami obliczeniowymi które uruchamiane są na serwerach obliczeniowych PLGrid. Z tego względu uruchomienie aplikacji Chipster możliwe jest na każdym komputerze wyposażonym w przeglądarkę internetową oraz środowisko Java Web Start. Usługa dostępna jest poprzez portal MBDAT bądź pod adresem: http://chipster.biologia.plgrid.pl. Na wskazanej stronie należy wcisnąć link launch Chipster. Alternatywnie, można również wybrać wersję aplikacji klienckiej możliwą do zastosowania na komputerach o większej ilości pamięci RAM: 3 lub 6 GB. Po wciśnięciu linku pobrany zostanie plik startowy aplikacji chipster.jnlp . Pobrany plik należy uruchomić za pomocą Java Web Start. Podczas uruchamiania aplikacji klienckiej nastąpi monit o zalogowanie z użyciem danych konta PLGrid.
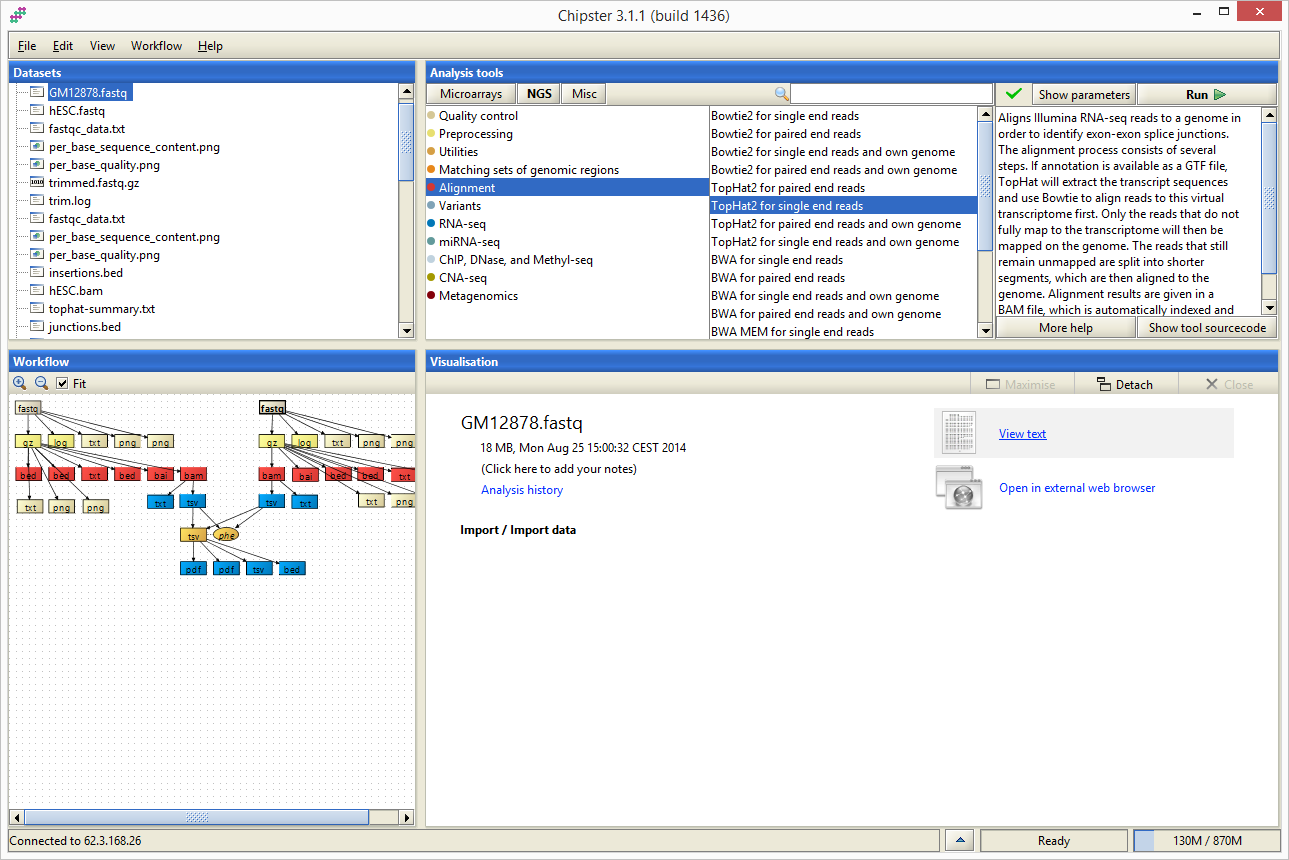
Organizacja interfejsu
Interfejs aplikacji Chipster podzielony jest na 4 obszary: Datasets , Workflow , Analysis tools oraz Visualisation . W obszarach Datasets oraz Workflow możliwe jest śledzenie wyników analiz oraz wskazywanie plików, na których mają być uruchomione narzędzia dostępne w obszarze Analysis tools . Ostatni z obszarów, Visualisation , służy do wyświetlania informacji o wybranym pliku/wyniku oraz do wyświetlania jego wizualizacji.
...
- Aby zapisać sesję na serwerze, w dowolnym momencie należy z menu File wybrać opcję Save Cloud Session.
- Po zapisaniu sesji można zamknąć aplikacje kliencką.
- Aby powrócić do zapisanej sesji należy po uruchomieniu wybrać z manu File opcję Open Cloud Session. Po wybraniu odpowiedniego pliku spowoduje to załadowanie wcześniej zapisanej sesji.
- Jeśli sesja zostanie zapisana w momencie kiedy aktywne są obliczenia (Running Jobs w prawym rogu dolnego paska stanu aplikacji), pozostaną one nadal aktywne, a ich wyniki po ukończeniu będą widoczne po ponownym załadowaniu zapisanej sesji. Opcja ta jest zalecana, kiedy zlecane są długotrwałe obliczenia.
- Ilość przestrzeni dyskowej na zapisane sesje jest ograniczona. Prosimy o rozsądne gospodarowanie miejscem. Niepotrzebne sesje można usuwać za pomocą File -> Manage Cloud Sessions.
- Przestrzeń dyskowa dostępna w ramach sesji zdalnych nie posiada kopii zapasowej! Zalecamy tworzenie kopii istotnych danych poprzez lokalne zachowywanie sesji lub poszczególnych plików na komputerze użytkownika.
...
Przykładowy scenariusz użycia: analiza danych RNA-seq
- Pobierz, a następnie załaduj do Chipster (używając polecenia File -> Import files ) pliki :
adrenal_1.fastq
adrenal_2.fastq
brain_1.fastq
brain_2.fastq
chr19_hg19.bed
chr19_iGenomes_GRCh37.gtf - Mapowanie odczytów do genomu referencyjnego.Narzędzie: Alignment -> Bowtie2 for paired end reads . Przed wyborem narzędzia i parametrów zaznacz pliki adrenal_1.fastq oraz adrenal_2.fastq . Wybieramy narzędzie i parametry:
- Genome: Homo_sapiens.GRch37.75
- No 1 mate reads: plik zawierający odczyty „forward” (zazwyczaj z ‘_1’ albo ‘f’ w nazwie): adrenal_1.fastq
- No 2 mate reads: plik zawierający odczyty „reverse” (zazwyczaj z ‘_2’ albo ‘r’ w nazwie): adrenal_2.fastq
Powtórz procedurę dla plików brain_1.fastq i brain_2.fastq . Przejrzyj dostępne wizualizacje plików wynikowych. W wizualizacji genome browser należy wybrać ten sam genom, który był użyty do mapowania. Dane są dostępne dla rejonu: Chr19:3000000:3500000.
- Analiza jakości mapowania. Narzędzie: Quality Control -> RNA-seq quality metrics with RseQC . Narzędzie należy uruchomić na pliku BAM uzyskanym z mapowania w poprzednim kroku oraz załadowanym pliku chr19_hg19.bed . Przeanalizuj otrzymane wykresy oraz informacje diagnostyczne.
- Zliczanie ilości odczytów zmapowanych w obrębie genów. Narzędzie RNA-seq -> Count aligned reads per genes with HTseq-count . Uruchamiamy dla plików bam uzyskanych z mapowania paired-end z użyciem bowtie2. Parametry:
- Reference organism: Homo_sapiens.GRch37.75
- Does the BAM file contain paired-end data: yes
- Was a data produced with a strand-specific protocol: yes
- Zdefiniowanie układu eksperymentalnego. Narzędzie: Utilities -> Define NGS experiment . Uruchamiamy na plikach htseq-count.tsv z poprzedniego kroku (zaznaczyć oba przed uruchomieniem). W opcjach należy zaznaczyć kolumnę zawierającą zliczenia w obrębie plików wybranych do analizy, w naszym przypadku „count”. Po uruchomieniu powstaje tabela łącząca zliczenia z obu plików oraz plik phenodata.tsv . Plik ten należy zaznaczyć i w oknie wizualizacji wybrać Phenodata editor . Teraz należy przyporządkować próbki do grup eksperymentalnych. W naszym przypadku nie mamy replikatów, więc sample001.tsv opisujemy w kolumnie „group” jako „adrenal”, natomiast sample0002.tsv jako „brain” i naciskamy „close”.
- Testowanie statystyczne genów pod względem różnicowej ekspresji. Narzędzie: RNA-seq -> Differential expression using edgeR . Narzędzie uruchom na plikach otrzymanych w poprzednim kroku. Parametry pozostaw domyślne. Przeanalizuj wykresy diagnostyczne oraz listę genów ulegających statystycznie istotnej różnicowej ekspresji.
Gdzie szukać dalszych informacji?
Szczegółowe informacje o użytkowaniu infrastruktury PLGrid znajdują się w Podręczniku Użytkownika.
...