Usługa jest przeznaczona dla biologów oraz bioinformatyków.
Chipster jest implementacją popularnego środowiska zarządzania zadaniami pozwalającego na uproszczone uruchamianie analiz bioinformatycznych na zasobach obliczeniowych PLGrid. W ramach usługi szczególny nacisk położony został na udostępnienie możliwie dużej liczby narzędzi związanych z analizą danych pochodzących z eksperymentów opartych o metody wysokoprzepustowego sekwencjonowania. Wykonywanie analiz z użyciem Chipster oparte jest o wygodny interfejs dostępny w formie aplikacji Java, pozwalający na intuicyjne zarządzanie danymi, narzędziami oraz wynikami. Wbudowane moduły wizualizacji pozwalają na przejrzystą i efektywną analizę wyników.
Aby skorzystać z usługi Chipster, należy mieć aktywne konto w infrastrukturze PLGrid.
Do usługi Chipster może uzyskać dostęp każdy użytkownik PLGrid, który jest użytkownikiem usługi Molecular Biology Data Analysis Toolkit. W celu uzyskania dostępu do tych usług należy wejść na stronę http://portal.plgrid.pl a następnie zalogować się podając swój identyfikator plgrid (np. plgkowalski) i hasło do portalu, po czym z górnej belki zawierającej menu wybrać opcję „Moje konto”. W prawej kolumnie ukaże się Katalog usług dostępnych dla danego użytkownika.
Z katalogu usług należy wybrać kategorię: Platforma Dziedzinowa – Biologia, a następnie „rozwiń”, ukaże się lista usług w tej kategorii, wśród nich będzie usługa o nazwie Molecular Biology Data Analysis Toolkit a tuż obok link zatytułowany „aplikuj o usługę”. Akceptacja użytkownika następuje automatycznie. Gdy rejestracja się powiedzie, usługa na liście w katalogu zyska status „aktywny”.
Uruchomienie usługi
Uruchomienie aplikacji Chipster możliwe jest na każdym komputerze wyposażonym w przeglądarkę internetową oraz środowisko Java Web Start. Usługa dostępna jest poprzez portal MBDAT bądź pod adresem: http://chipster.biologia.plgrid.pl. Na wskazanej stronie należy wcisnąć link launch Chipster. Alternatywnie, można również wybrać wersję aplikacji możliwą do zastosowania na komputerach o większej ilości pamięci RAM: 3 lub 6 GB. Po wciśnięciu linku pobrany zostanie plik startowy aplikacji chipster.jnlp. Pobrany plik należy uruchomić za pomocą Java Web Start. Podczas uruchamiania aplikacji nastąpi monit o zalogowanie z użyciem danych konta PLGrid.
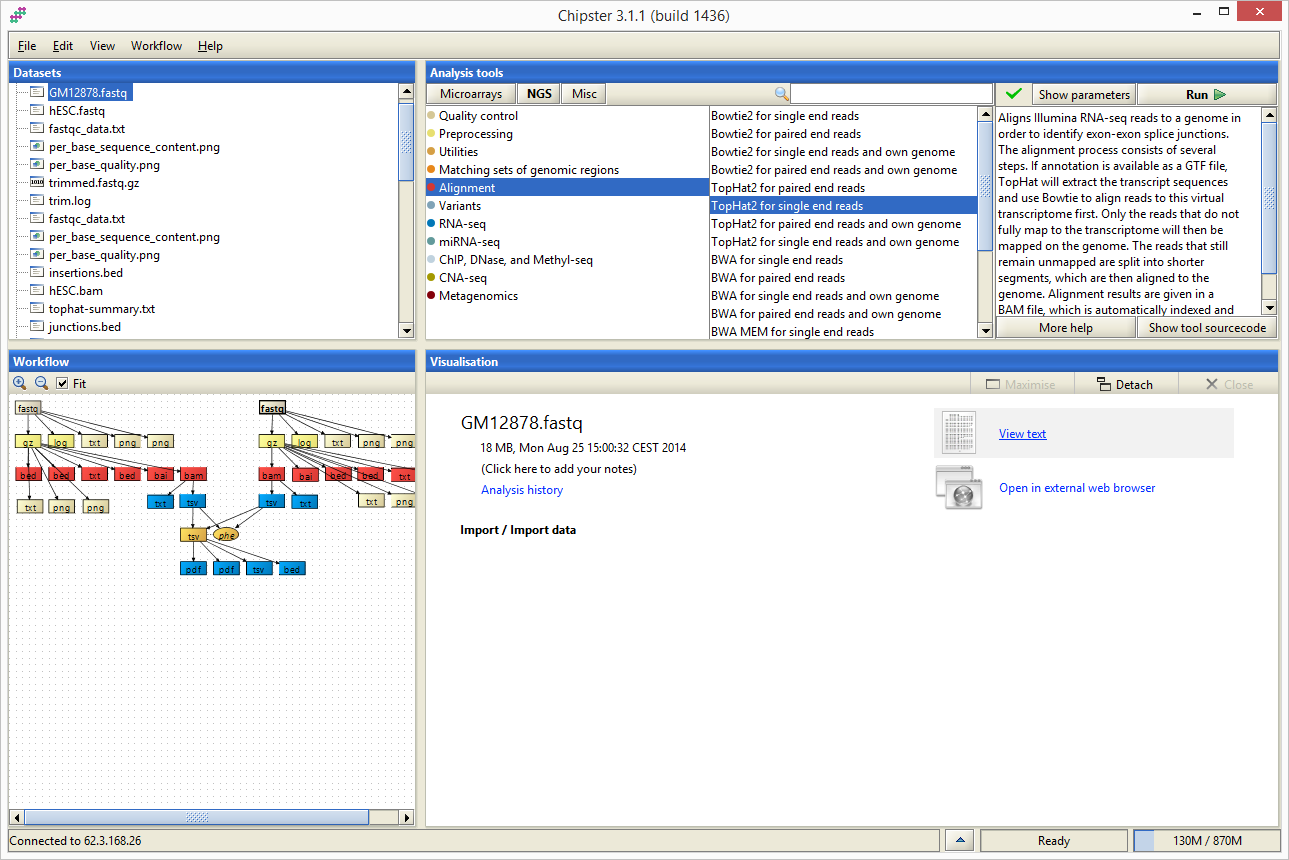
Organizacja interfejsu

Interfejs aplikacji Chipster podzielony jest na 4 obszary: Datasets, Workflow, Analysis tools oraz Visualisation. W obszarach Datasets oraz Workflow możliwe jest śledzenie wyników analiz oraz wskazywanie plików na których mają byc uruchomione narzędzia dostepne w obszarze Analysis tools. Ostatni z obszarów, Visualisation, służy do wyświetlanai informacji o wybranym pliku/wyniku oraz do wyświetlania jego wizualizacji.
Przykładowy scenariusz użycia: analiza danych RNA-seq
Szczegółowe informacje o użytkowaniu infrastruktury PLGrid znajdują się w Podręczniku Użytkownika.
Szczegółowa dokumentacja Chipster wraz z przykładowymi sesjami znajduje się na stronie: http://chipster.csc.fi/manual/
Informacje o usługach dziedzinowych Biologia dostępne są na stronie: http://biologia.plgrid.pl/
Uzyskanie informacji/helpdesk PLGrid: dokumentacji o pomocy