Page History
...
Z usługi można korzystać na dwa sposoby: za pośrednictwem interaktywnego portalu UNRES oraz przy pomocy systemy wsadowego QosCos QosCosGrid.
Aktywowanie usługi
Aby skorzystać z usługi UNRES należy posiadać konto w portalu PLGRID oraz posiadać aktywny grant obliczeniowywłaściwy PLGrid. Należy również złożyć wniosek o dołączenie usługę UNRES w Katalogu Aplikacji i Usług. Spowoduje to dodanie użytkownika do zespołu plggunresng (zespół użytkowników usługi UNRES).
...
Obecnie aplikacje składające się na usługę są zainstalowane w ramach infrastruktury PLGrid tylko na klastrze inula na klastrach:
- Inula w Poznańskim Centrum Superkomputerowo-Sieciowym
- Prometheus w Cyfronecie.
Portal QCG umożliwiający interaktywne uruchamianie zadań kieruje zadania tylko na klaster inula. Za pośrednictwem portalu QCG można uruchomić tylko główny moduł obliczeniowy UNRES, natomiast moduły analizy wyników dynamiki molekularnej z wymianą replik można uruchomić jedynie bezpośrednio w trybie wsadowym. Rozszerzenie funkcjonalności portalu QCG na ten moduły jest w toku.
Pierwsze kroki
Ponieważ w chwili obecnej system QosCosGrid nie obejmuje kierowania zadań na klaster Prometheus, w przypadku uruchamiania zadań na tym komputerze należy używać systemu slurm, podobnie jak w przypadku uruchamiania innych zadań na tym komputerze. Szczegółowy opis znajduje się w podręczniku Prometheus:Podstawy.
Pierwsze kroki
Zalecane jest zapoznanie się z dokumentacją części składowych pakietu UNRES dostępną na stronie pakietu (dokumentacja w języku Zalecane jest zapoznanie się z dokumentacją części składowych pakietu UNRES dostępną na stronie pakietu (dokumentacja w języku angielskim).
Uruchamianie przez portal UNRES
Zlecanie zadań przez portal UNRES możliwe jest tylko na klaster inula w PCSS.
Portal UNRES jest dostępny pod adresem https://unres-portal.plgrid.pl. Portal jest zintegrowany z OPENID. Należy zalogować się na portal używając identyfikatora użytkownika w systemie PL-GRID.
...
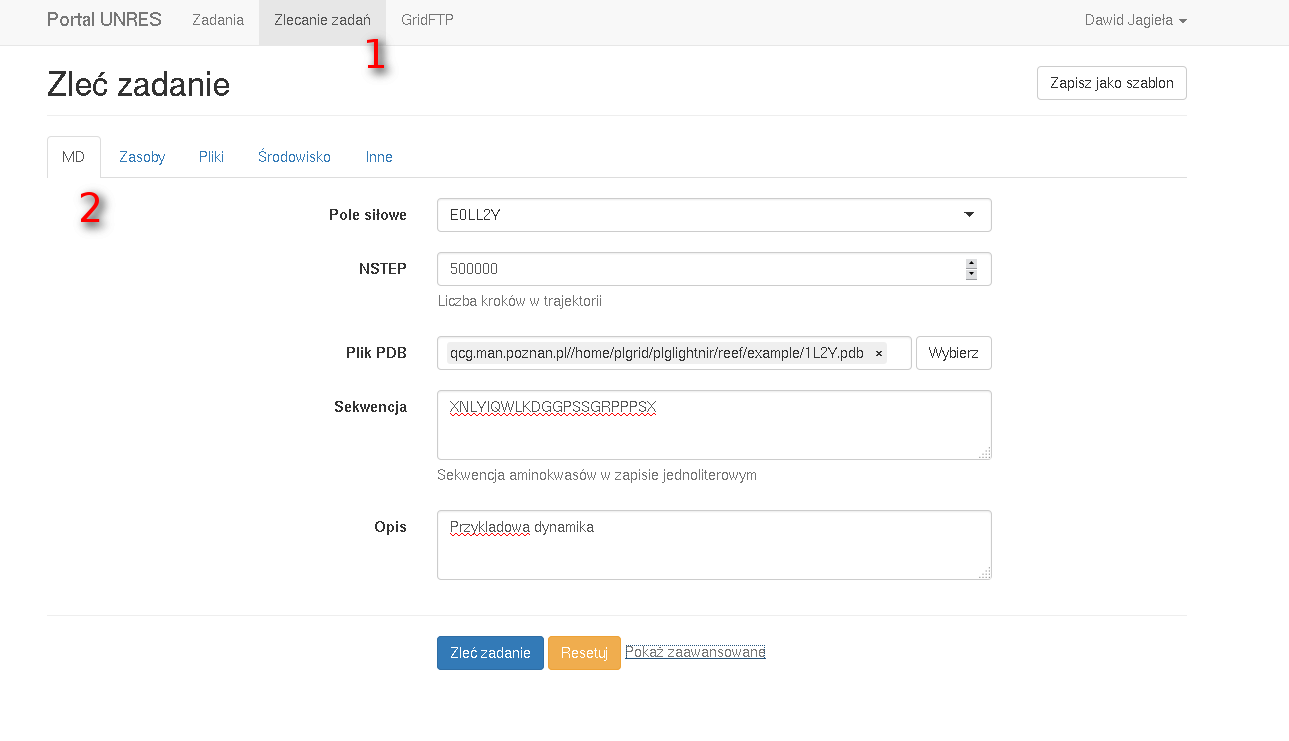
- ustawić pole siłowe (GAB lub E0LL2Y)
- ustawić liczbę kroków dynamiki
- wybrać plik PDB będący strukturą referencyjną
- podać sekwencję aminokwasów w pliku PDB w zapisie jednoliterowych uwzględniając aminokwasy terminalne w zapisie pakietu UNRES.
Uwaga 1: Wybranie pliku pdb, który zawiera strukturę odniesienia danego białka (na ogół strukturę eksperymentalną z bazy pdb), jest o w obecnej konfiguracji portalu konieczne. Jeżeli ten plik nie zostanie podany, zadanie zakończy się z błędem.
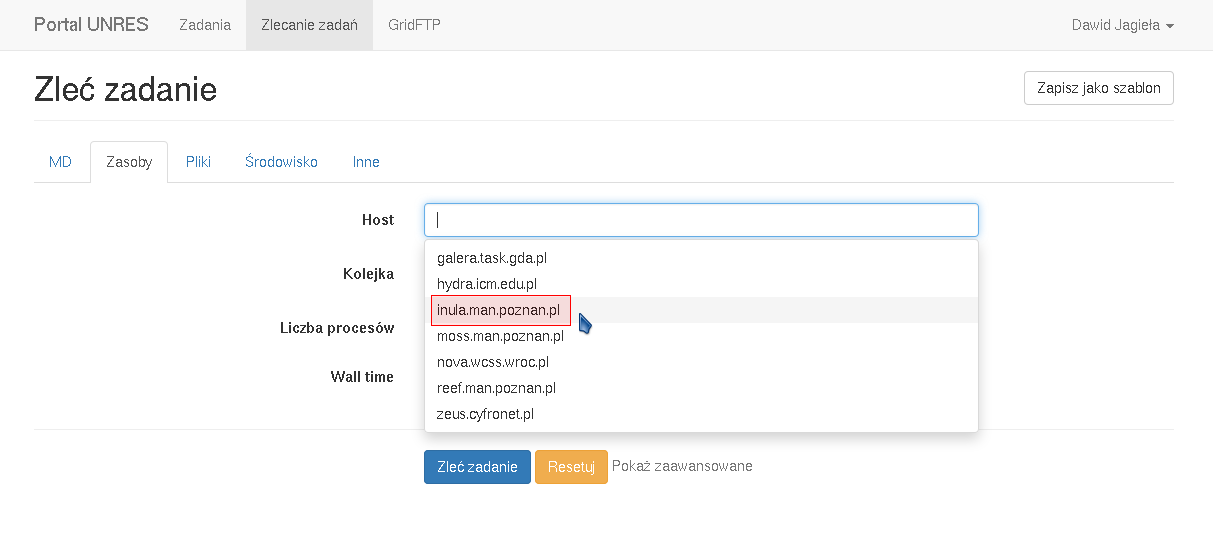
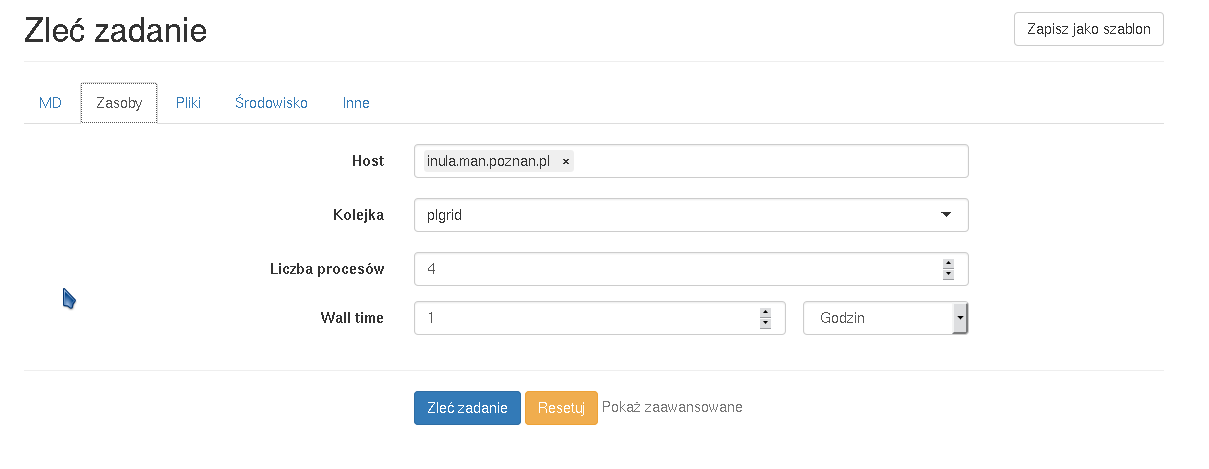
Następnie w zakładce "Zasoby" ustawiamy klaster obliczeniowy oraz czas obliczeń. UWAGA: W chwili obecnej pakiet UNRES zaimplementowany jest tylko na klastrze Inula.
Więcej opcji dynamiki można uzyskać po kliknięciu napisu "Zaawansowane" w zakładce MD. Odpowiednie pola są objaśnione poniżej; wartości domyślne podano w nawiasach:
NTWE (100): częstość zapisu energii w dynamice molekularnej.
NTWX (1000): częstść zapisu współrzędnych cząsteczki białka.
DT: (0.1) długość kroku czasowego; jednostka jest równa 48,9 fs.
Aby wybrać grant w ramach którego będą wykonywane obliczenia, należy wykonać następujące czynności:
1. wybrać zakładkę MD,
2. kliknąć napis "pokaż zaawansowane",
3. wpisać w pole Grant ID grantu.
Program UNRES wygeneruje tyle niezależnych trajektorii ile wynosi liczba procesorów.
Plikami wynikowymi, które po poprawnym zakończeniu zadania pojawią się w katalogu roboczym i będą mogły być pobrane przez gftp, są:
ZADANIE.out_GB000 - ZADANIE.out_GB00n (gdzie zadanie jest prefiksem nadawanym przez portal a n+1 jest liczbą procesorów): główne pliki wynikowe. Zawierają podstawowe informacje o układzie, informacje o ustawieniach obliczeń dynamiki molekularnej oraz ewentualne informacje o błędach w danych.
ZADANIE_GB000.stat - ZADANIE_GB00n : pliki zawierające podstawowe informacje o przebiegu dynamiki dla każdej trajektorii (nr kroku, czas, energia, rmsd od struktury odniesienia). Proszę zapoznać się z opisem plików danych i wynikowych programu UNRES (dokumentacja w języku angielskim).
ZADANIE_MD000.pdb - ZADANIE_MD00n.pdb : zrzuty konformacji w formacje pdb dla każdej trajektorii.
Uruchamianie bezpośrednio w systemie wsadowym
W celu zdefiniowania ścieżek dostępu oraz ustawienia wartości zmiennych środowiskowych dla usługi należy załadować moduł plgrid/apps/unres-e0ll2y/3.2.1 (pole E0LL2Y0) lub plgrid/apps/unres-gab/3.2.1 (pole GAB), w zależności od żądanego pola siłowego. W przypadku uruchamiania zadań przez portal odpowiedni moduł jest ładowany poprzez wybór odpowiedniego pola siłowego z menu.
Przykłady przygotowywania plików wsadowych oraz danych do uruchamiania usługi UNRES w różnych wariantach wraz z opisem zawarte są w archiwum zip. przykłady.zip można pobrać korzystając z przytoczonego linku.
Poniżej krótki opis wraz z objaśnieniem uruchamiania kanonicznej dynamiki molekularnej Langevina. Używany jest wariant pola siłowego UNRES oznaczony jako pole E0lL2Y (pole "ogólnych zastosowań"). Ten przykład jest zawarty w przytoczonym archiwum w katalogu ff_1l2y_1le1/LANG.
Uwaga 2: Sekwencja aminokwasowa musi zaczynać/kończyć się resztą "dummy" oznaczaną jako X jeżeli łańcuch nie zaczyna się/kończy pełną grupą peptydową (czyli grupa początkowa jest grupą -NH3+ lub grupa końcowa jest grupą COO-; sekwencja "nieblokowana"). Jeżeli łańcuch zaczyna się/kończy pełną grupą peptydową lub pierwszą/ostatnią resztą jest glicyna, należy wstawić G. Wstawienie innych reszt niź X lub G na początku lub końcu spowoduje błędy w obliczeniach. Wstawienie grup blokujących powoduje pozorne wydłużenie sekwencji, np. jeżeli chcemy wykonać obliczenia nieblokowanego pentapeptydu Ala-Lys-Gly-Pro-Ser to musimy wprowadzić sekwencję XAKGPSX a jeżeli chcemy wykonać obliczenia analogicznego blokowanego pentapeptydu Ac-Ala-Lys-Gly-Pro-Ser-NHCH3 to wprowadzamy sekwencję GAKGPSG. W przypadku sekwencji np. Gly-Leu-Pro-Leu-Ala-Gly nie trzeba dodawać grup blokujących i wprowadzić sekwencję GLPLAG
Następnie w zakładce "Zasoby" ustawiamy klaster obliczeniowy oraz czas obliczeń. UWAGA: W chwili obecnej portal uruchamia zadania jedynie na klastrze inula).
Więcej opcji dynamiki można uzyskać po kliknięciu napisu "Zaawansowane" w zakładce MD. Odpowiednie pola są objaśnione poniżej; wartości domyślne podano w nawiasach:
NTWE (100): częstość zapisu energii w dynamice molekularnej.
NTWX (1000): częstść zapisu współrzędnych cząsteczki białka.
DT: (0.1) długość kroku czasowego; jednostka jest równa 48,9 fs.
Aby wybrać grant w ramach którego będą wykonywane obliczenia, należy wykonać następujące czynności:
1. wybrać zakładkę MD,
2. kliknąć napis "pokaż zaawansowane",
3. wpisać w pole Grant ID grantu.
Program UNRES wygeneruje tyle niezależnych trajektorii ile wynosi liczba procesorów.
Plikami wynikowymi, które po poprawnym zakończeniu zadania pojawią się w katalogu roboczym i będą mogły być pobrane przez gftp, są:
ZADANIE.out_GB000 - ZADANIE.out_GB00n (gdzie zadanie jest prefiksem nadawanym przez portal a n+1 jest liczbą procesorów): główne pliki wynikowe. Zawierają podstawowe informacje o układzie, informacje o ustawieniach obliczeń dynamiki molekularnej oraz ewentualne informacje o błędach w danych.
ZADANIE_GB000.stat - ZADANIE_GB00n : pliki zawierające podstawowe informacje o przebiegu dynamiki dla każdej trajektorii (nr kroku, czas, energia, rmsd od struktury odniesienia). Proszę zapoznać się z opisem plików danych i wynikowych programu UNRES (dokumentacja w języku angielskim).
ZADANIE_MD000.pdb - ZADANIE_MD00n.pdb : zrzuty konformacji w formacje pdb dla każdej trajektorii. Pliki te można bezpośrednio oglądać przeglądarkami do molekuł np. pymol.
Uruchamianie bezpośrednio w systemie wsadowym
Uruchamianie zadań na klastrze inula
W celu zdefiniowania ścieżek dostępu oraz ustawienia wartości zmiennych środowiskowych dla usługi należy załadować moduł plgrid/apps/unres-e0ll2y/3.2.1 (pole E0LL2Y0) lub plgrid/apps/unres-gab/3.2.1 (pole GAB), w zależności od żądanego pola siłowego. W przypadku uruchamiania zadań przez portal odpowiedni moduł jest ładowany poprzez wybór odpowiedniego pola siłowego z menu.
Przykłady przygotowywania plików wsadowych oraz danych do uruchamiania usługi UNRES w różnych wariantach wraz z opisem zawarte są w archiwum zip. przykłady.zip można pobrać korzystając z przytoczonego linku.
Poniżej krótki opis wraz z objaśnieniem uruchamiania kanonicznej dynamiki molekularnej Langevina. Używany jest wariant pola siłowego UNRES oznaczony jako pole E0lL2Y (pole "ogólnych zastosowań"). Ten przykład jest zawarty w przytoczonym archiwum w katalogu ff_1l2y_1le1/LANG.
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
Skrypt QCG (plik langdyn.qcg; nie ma w nim dodanych tutaj komentarzy):
-------------------------------------------------------------------------------------
Skrypt QCG (plik langdyn.qcg; nie ma w nim dodanych tutaj komentarzy):
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
#QCG host=inula #QCG host=inula
## Obliczenia na inuli (na razie
## Obliczenia na inuli (na razie jedyna możliwość)
#QCG walltime=PT15M
## maksymalny czas zadania 15 minut
#QCG nodes=1:1
## używamy 1 procesor
#QCG note=l2y-langdyn
## identyfikator zadania
#QCG output=${JOB_ID}.output
## specyfikacja nazw pliku z wydrukami z programu i systemu qcg na ekran
#QCG error=${JOB_ID}.error
## specyfikacja nazwy pliku z komunikatami o błędach
#QCG stage-in-dir=.
## pliki danych będą brane z bieżącego katalogu
#QCG stage-out-dir=.
## pliki wynikowe będą umieszczane w bieżącym kagalogu
module load plgrid/apps/unres-e0ll2y/3.2.1
## ładowanie modułu zawierającego komp0onentu usługi dla pola E0LL2Y
export NPROCS=`cat $PBS_NODEFILE | wc -l`
cd $PBS_O_WORKDIR
mpirun -machinefile $PBS_NODEFILE -np $NPROCS unres.csh
## uruchamiamie skryptu unres.csh
...
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
Uruchamianie
Po zalogowaniu się na serwer, z którego można uruchamiać zadania w systemie Qcg-Qoscos, np. qcg.man.poznan.pl i przeniesieniu tam wymienionych powyżej plików, należy wydać polecenie:
...
Plikami wynikowymi będą:
1L2Y_lang,.out_MD000GB000 : plik zawierający podstawowe informacje o danych wejściowych, typie obliczeń, zastosowanych ustawieniach oraz przebiegu obliczeń.
...
Trajektoria zapisana w formie skompresowanej w pliku cx nie jest gotowa do oglądania/przetwarzania programami grafiki molekularnej. Należy przekształcić go do formatu Protein Data Bank (PDB) przy użyciu programu xdrf2pdb. Program jest dostępny w ramach usługi UNRES i może być uruchamiany w trybie wsadowym jednak z uwagi na to, że pliki pdb zajmują znacznie więcej miejsca niż pliki cx, lepiej skopiować plik cx na swój lokalny komputer i tam uruchomić xdrf2pdb interaktywnie. Program xdrf2pdb można uzyskać ze strony "downloads" pakietu UNRES. Po wejściu na stronę należy kliknąć na guzik "ACCEPT LICENSE TERMS" i ściągnąć archiwum xdrfpdb-src-v.3.2.1.tar.gz.
Polecenie konwersji dla podanego przykładu jest następujące:
xdrf2pdb three seq-3let 1L2Y_lang_MD000.cx 1 1 100 1L2Y_lang.pdbgdzie
three oznacza, że podawana będzie sekwencja aminokwasowa białka w kodzie trójliterowym
seq-3let jest plikiem z sekwencją; format 20(A3, 1X); zawartość poniżej
D ASN LEU TYR ILE GLN TRP LEU LYS ASP GLY GLY PRO SER SER GLY ARG PRO PRO PROSER D1L2Y_lang_MD000.cx jest nazwą pliku ze współrzędnymi konformacji białka z obliczonej trajektorii w formacie skompresowanym.
1 1 100 : konformacje będą zapisywane co 1, począwszy od konformacji 1 a skończywszy na konformacji 100
"downloads" pakietu UNRES. Po wejściu na stronę należy kliknąć na guzik "ACCEPT LICENSE TERMS" i ściągnąć archiwum xdrfpdb-src-v.3.2.1.tar.gz. Program xdrf2pdb wymaga biblioteki xdrf, która jest w pakiecie UNRES. Jeżeli jeszcze jej nie posiadamy to trzeba ściągnąć archiwum lib.tar.gz ze strony pakietu UNRES. Można też ściągnąć cały pakiet UNRES unrespack-v.3.2.1.tar.gz; biblioteka jest w sources/lib/xdrf lub xdrf_em64 (64 bit) albo xdrf-Argonne (BlueGene Q) gdyby ta nie zadziałała.
Polecenie konwersji dla podanego przykładu jest następujące:
xdrf2pdb three seq-3let 1L2Y_lang_MD000.cx 1 1 100 1L2Y_lang.pdbgdzie
three oznacza, że podawana będzie sekwencja aminokwasowa białka w kodzie trójliterowym
seq-3let jest plikiem z sekwencją; format 20(A3, 1X); zawartość poniżej
D ASN LEU TYR ILE GLN TRP LEU LYS ASP GLY GLY PRO SER SER GLY ARG PRO PRO PROSER D1L2Y_lang_MD000.cx jest nazwą pliku ze współrzędnymi konformacji białka z obliczonej trajektorii w formacie skompresowanym.
1 1 100 : konformacje będą zapisywane co 1, począwszy od konformacji 1 a skończywszy na konformacji 100
1L2Y_lang.pdb : wynikowy plik z konformacjami w formacie PDB; konformacje te zawierają geometrię na poziomie gruboziarnistym; tylko atomy węgla alfa (CA) i centra łańcuchów bocznych (CB).
Uruchamianie zadań na klastrze Prometheus
Zadania są uruchamiane w systemie kolejkowania slurm. W zależności od używanego pola należy załadować jeden z modułów:
- plgrid/apps/unres-e0ll2y/3.2.1 (w tym przypadku należy uruchomić aplikację unresMD_ifort_MPI_E0LL2Y.exe lub odpowiedni stowarzyszony z nią program przetwarzający wyniki symulacji)
- plgrid/apps/unres-gab/3.2.1 (w tym przypadku należy uruchomić aplikację unresMD_ifort_MPI_GAB.exe lub odpowiedni stowarzyszony z nią program przetwarzający wyniki symulacji)
Dodatkowo należy załadować moduł plgrid/tools/impi.
Podobnie jak w przypadku klastra inula, załadowanie modułów powoduje zdefiniowanie plików z właściwymi dla danego pola parametrami; pliki te można jednk podmienić na pliki z parametrami użytkownika. Przykładowy skrypt jest podany poniżej.
----------------------------------------------------------
Plik energy-pdb.sbatch
----------------------------------------------------------
#!/bin/bash -l
#SBATCH -J unres
#SBATCH -N 1
#SBATCH --ntasks-per-node=1
#SBATCH -n 1
#SBATCH --time=00:01:00
#SBATCH -A unresprometheustest
#SBATCH -p plgrid
#SBATCH --output="output.out"
#SBATCH --error="error.err"
#
module load plgrid/apps/unres-e0ll2y/3.2.1
module add plgrid/tools/impi
export PREFIX=1L2Y_ene-pdb
export OUT1FILE=YES
export FGPROCS=1
mpirun unresMD_ifort_MPI_E0LL2Y.exe
-----------------------------------------------------------------
Przykładowe pliki wsadowe dla systemu slurm oraz pliki danych i pliki wynikowe dla różnych trybów obliczeń wraz z krótkim opisem są dostępne w katalogu /net/software/local/unres/Przyklady na klastrze Prometheus; archiwum można pobrać stąd w postaci pliku Przyklady_prometheus.zip1L2Y_lang.pdb : wynikowy plik z konformacjami w formacie PDB; konformacje te zawierają geometrię na poziomie gruboziarnistym; tylko atomy węgla alfa (CA) i centra łańcuchów bocznych (CB).
Zaawansowane użycie
Przewidywanie de novo struktury białka przy pomocy pola siłowego UNRES
...
Strona dokumentacji pakietu UNRES ze szczegółowym opisem wprowadzanych danych (w języku angielskim).