Page History
...
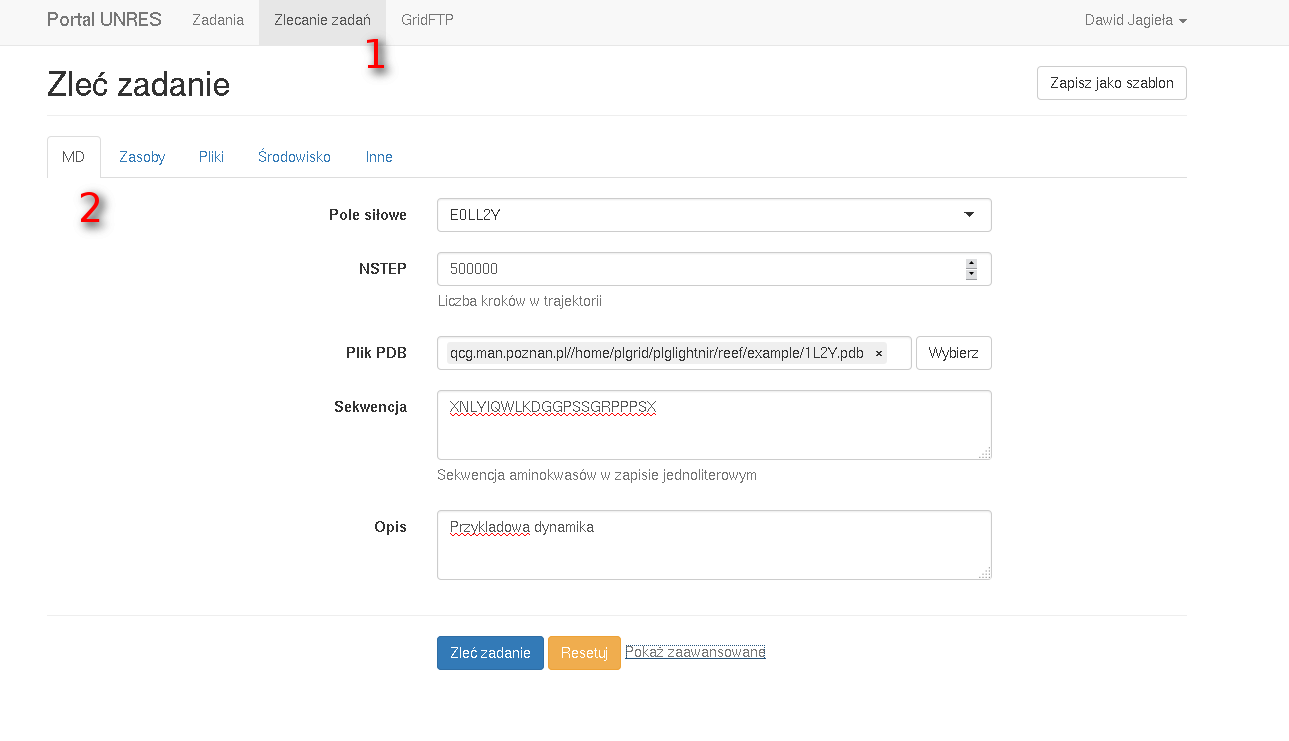
- ustawić pole siłowe (GAB lub E0LL2Y)
- ustawić liczbę kroków dynamiki
- wybrać plik PDB będący strukturą referencyjną
- podać sekwencję aminokwasów w pliku PDB w zapisie jednoliterowych uwzględniając aminokwasy terminalne w zapisie pakietu UNRES.
Uwaga: Wybranie pliku pdb, który zawiera strukturę odniesienia danego białka (na ogół strukturę eksperymentalną z bazy pdb), jest o w obecnej konfiguracji portalu konieczne. Jeżeli ten plik nie zostanie podany, zadanie zakończy się z błędem.
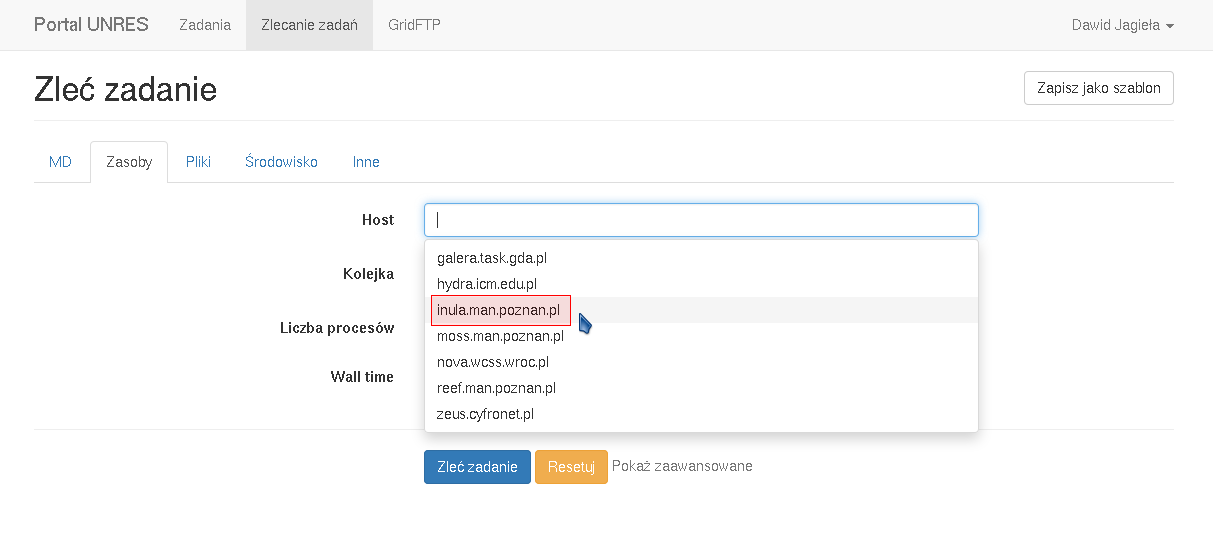
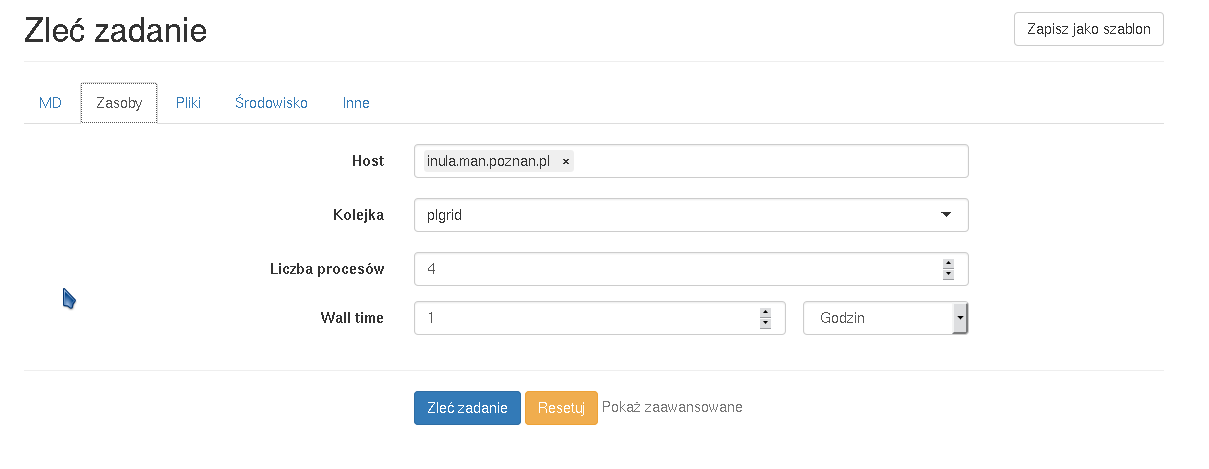
Następnie w zakładce "Zasoby" ustawiamy klaster obliczeniowy oraz czas obliczeń. UWAGA: W chwili obecnej pakiet UNRES zaimplementowany jest tylko na klastrze Inula.
...
3. wpisać w pole Grant ID grantu.
Program UNRES wygeneruje tyle niezależnych trajektorii ile wynosi liczba procesorów.
Plikami wynikowymi, które po poprawnym zakończeniu zadania pojawią się w katalogu roboczym i będą mogły być pobrane przez gftp, są:
ZADANIE.out_GB000 - ZADANIE.out_GB00n (gdzie zadanie jest prefiksem nadawanym przez portal a n+1 jest liczbą procesorów): główne pliki wynikowe. Zawierają podstawowe informacje o układzie, informacje o ustawieniach obliczeń dynamiki molekularnej oraz ewentualne informacje o błędach w danych.
ZADANIE_GB000.stat - ZADANIE_GB00n : pliki zawierające podstawowe informacje o przebiegu dynamiki dla każdej trajektorii (nr kroku, czas, energia, rmsd od struktury odniesienia). Proszę zapoznać się z opisem plików danych i wynikowych programu UNRES (dokumentacja w języku angielskim).
ZADANIE_MD000.pdb - ZADANIE_MD00n.pdb : zrzuty konformacji w formacje pdb dla każdej trajektorii.
Uruchamianie bezpośrednio w systemie wsadowym
...