Page History
...
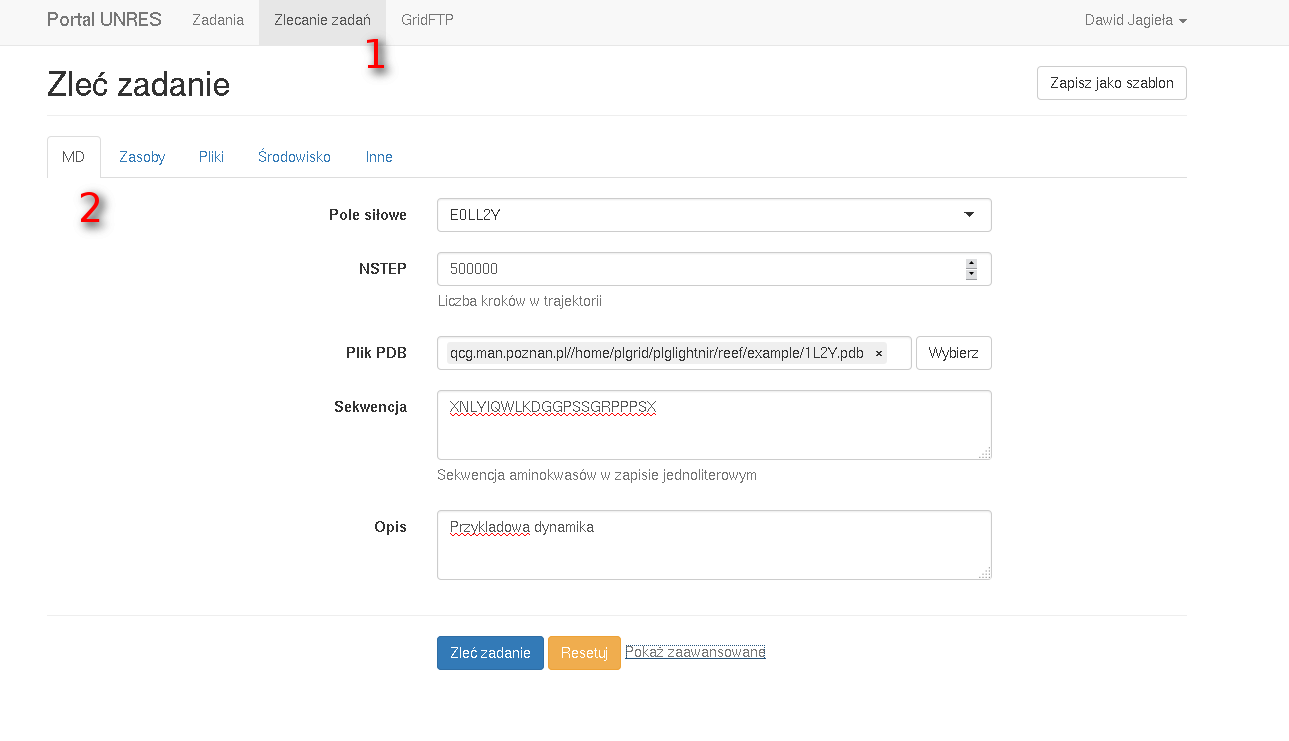
- ustawić pole siłowe (GAB lub E0LL2Y)
- ustawić liczbę kroków dynamiki
- wybrać plik PDB będący strukturą referencyjną
- podać sekwencję aminokwasów w pliku PDB w zapisie jednoliterowych uwzględniając aminokwasy terminalne w zapisie pakietu UNRES.
Uwaga 1: Wybranie pliku pdb, który zawiera strukturę odniesienia danego białka (na ogół strukturę eksperymentalną z bazy pdb), jest o w obecnej konfiguracji portalu konieczne. Jeżeli ten plik nie zostanie podany, zadanie zakończy się z błędem.strukturę eksperymentalną z bazy pdb), jest o w obecnej konfiguracji portalu konieczne. Jeżeli ten plik nie zostanie podany, zadanie zakończy się z błędem.
Uwaga 2: Sekwencja aminokwasowa musi zaczynać/kończyć się resztą "dummy" oznaczaną jako X jeżeli łańcuch nie zaczyna się/kończy pełną grupą peptydową (czyli grupa początkowa jest grupą -NH3+ lub grupa końcowa jest grupą COO-; sekwencja "nieblokowana"). Jeżeli łańcuch zaczyna się/kończy pełną grupą peptydową lub pierwszą/ostatnią resztą jest glicyna, należy wstawić G. Wstawienie innych reszt niź X lub G na początku lub końcu spowoduje błędy w obliczeniach. Wstawienie grup blokujących powoduje pozorne wydłużenie sekwencji, np. jeżeli chcemy wykonać obliczenia nieblokowanego pentapeptydu Ala-Lys-Gly-Pro-Ser to musimy wprowadzić sekwencję XAKGPSX a jeżeli chcemy wykonać obliczenia analogicznego blokowanego pentapeptydu Ac-Ala-Lys-Gly-Pro-Ser-NHCH3 to wprowadzamy sekwencję GAKGPSG. W przypadku sekwencji np. Gly-Leu-Pro-Leu-Ala-Gly nie trzeba dodawać grup blokujących i wprowadzić sekwencję GLPLAG
Następnie w zakładce "Zasoby" ustawiamy klaster obliczeniowy oraz czas obliczeń. UWAGA: W chwili obecnej pakiet UNRES zaimplementowany jest tylko na klastrze Inula.
...
ZADANIE_MD000.pdb - ZADANIE_MD00n.pdb : zrzuty konformacji w formacje pdb dla każdej trajektorii. Pliki te można bezpośrednio oglądać przeglądarkami do molekuł np. pymol.
Uruchamianie bezpośrednio w systemie wsadowym
...