Page History
...
Usługa UNRES jest przyjazną dla użytkownika realizacją pakietu modelowania gruboziarnistego białek, opartego na polu siłowym UNRES opracowanym w wyniku współpracy grupy prof. Adama Liwo z Wydziału Chemii Uniwersytetu Gdańskiego oraz grupy prof. Harolda A. Scheragi , z Cornell University, USA. Dzięki znacznemu uproszczeniu reprezentacji łańcucha polipeptydowego przy jednoczesnym ścisłym związku wyprowadzonego pola siłowego z fizyką oddziaływać symulacje są przyspieszone 1000 - 10000 razy w stosunku do obliczeń w reprezentacji pełnoatomowej. Adresatami usługi są biofizycy, biologowie molekularni i bioinformatycy, którzy mają potrzebę symulowania zainteresowani symulowaniem zmian strukturalnych, dynamiki i termodynamiki dużych układów białkowych w czasie rzeczywistym. W szczególności usługa będzie przydatna dla badaczy, którzy chcą wykonać wspomniane wielkoskalowe symulacje białek, natomiast brak czasu nie pozwala im na zapoznanie się z uruchamianiem zadań bezpośrednio na serwerach przy użyciu systemu kolejkowania.
...
- UNRES: właściwe symulacje,
- WHAM : przerwarzanie przetwarzanie wyników dynamiki molekularnej z wymianą replik metodą analizy ważonych histogramów,
- CLUSTER: analiza skupień wyników symulacji; analiza wyników dynamiki molekularnej z wymianą replik wymaga wstępnego przetworzenia metodą analizy ważonych histogramów.
...
Aby skorzystać z usługi UNRES należy posiadać konto w portalu PLGRID oraz posiadać aktywny grant obliczeniowy. Należy również złożyć wniosek o dołączenie do zespołu plggunresng zespołu plggunresng (zespół użytkowników usługi UNRES).
Ograniczenia w korzystaniu
...
Obecnie aplikacje składające się na usługę są zainstalowane w ramach systemu PLGRID infrastruktury PLGrid tylko na klastrze inula w Poznańskim Centrum Superkomputerowo-Sieciowym.
Obecnie za Za pośrednictwem portalu QCG można uruchomić tylko główny moduł obliczeniowy UNRES, natomiast moduły analizy wyników dynamiki molekularnej z wymianą replik można uruchomić jedynie bezpośrednio w trybie wsadowym. Rozszerzenie funkcjonalności portalu QCG na ten moduły jest w toku.
...
W celu zlecenia zadania z poziomu portalu UNRES należy wybrać zakładkę "Zlecanie zadań", a następnie przycisk "nowe zadanie":
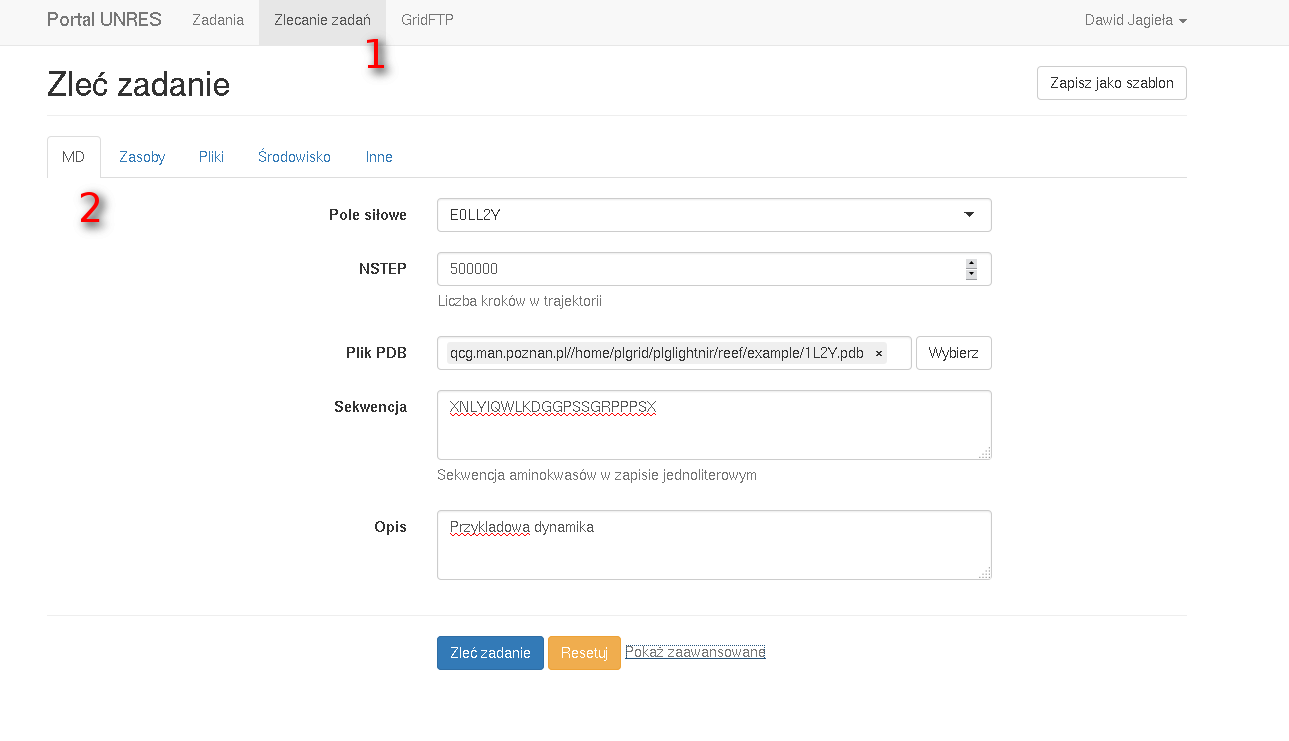
W celu zlecenia dynamiki molekularnej należy w zakładce "MD" :
- ustawić pole siłowe (GAB lub E0LL2Y)
- ustawić liczbę kroków dynamiki
- wybrać plik PDB będący strukturą referencyjną
- podać sekwencję aminokwasów w pliku PDB w zapisie jednoliterowych uwzględniając aminokwasy terminalne w zapisie pakietu UNRES.
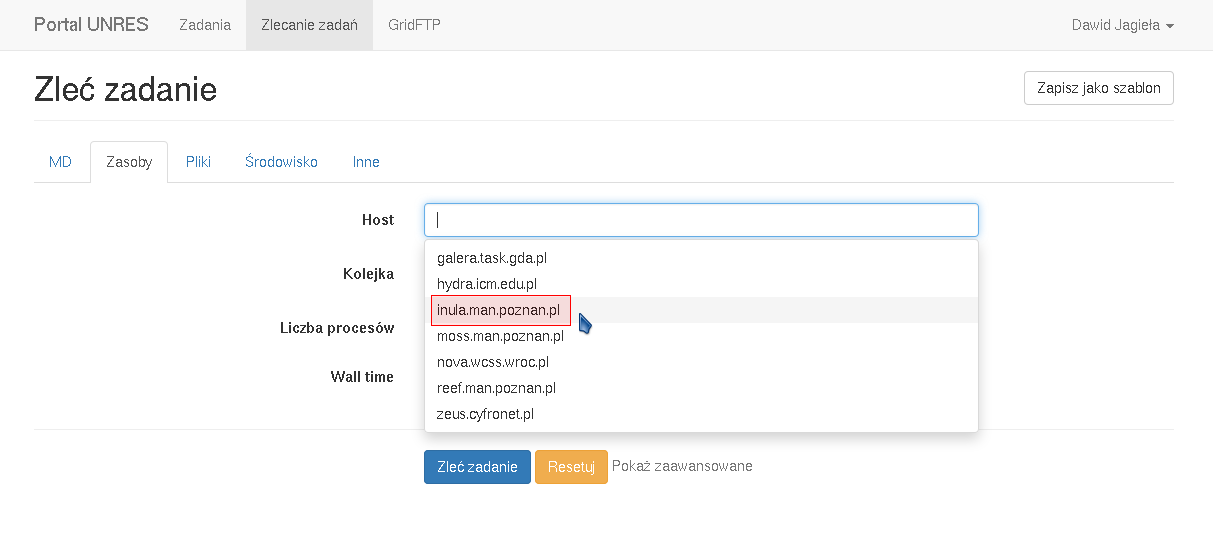
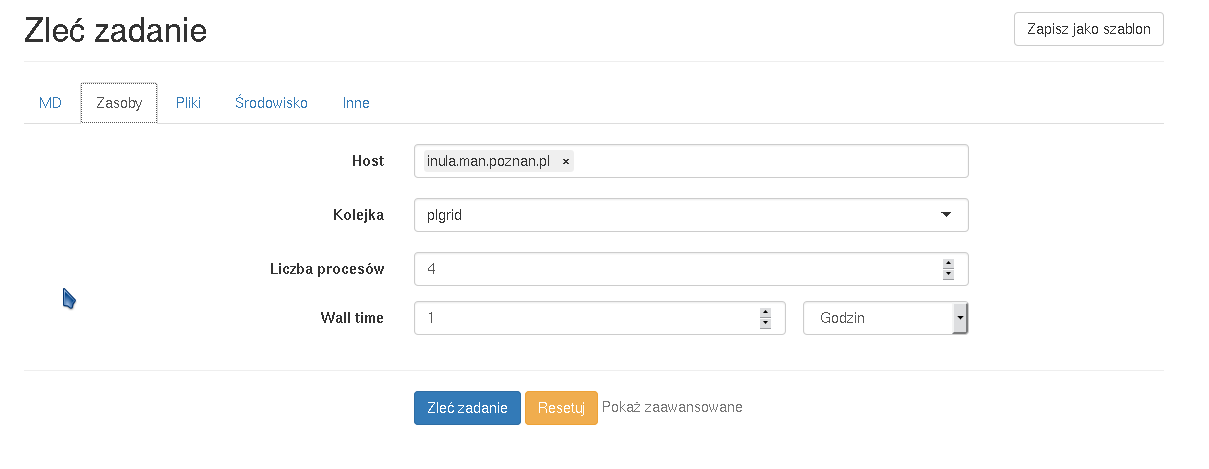
Następnie w zakładce "Zasoby" ustawiamy klaster obliczeniowy oraz czas obliczeń. UWAGA: W chwili obecnej pakiet UNRES zaimplementowany jest tylko na klastrze Inula.
Uruchamianie bezpośrednio w systemie wsadowym
Przykłady przygotowywania plików wsadowych oraz danych do uruchamiania usługi UNRES w różnych wariantach wraz z opisem zawaste zawarte są w archiwum zip. przykłady.zip można pobrać korzystając z przytoczonego linku.
Poniżej krótki opis wraz z objaśnieniem uruchamiania kanonicznej dynamiki molekularnej Langevina. Używany jest wariant pola siłowego UNRES oznaczony jako pole E0lL2Y (pole "ogólnych zastosowań"). Ten przykład jest zawarty w przytoczonym archiwum w katalogu ff_1l2y_1le1/LANG.
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Skrypt QCG (plik langdyn.qcg):
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------#QCG host=inula
...
# Obliczenia na inuli (na razie jedyna możliwość)
#QCG walltime=PT15M # maksymalny czas zadania 15 minut
#QCG nodes=1:1
...
# używamy 1 procesor
#QCG note=l2y-langdyn # identyfikator zadania
#QCG output=${JOB_ID}.output # specyfikacja nazw pliku z wydrukami z programu i systemu qcg na ekran
#QCG error=${JOB_ID}.error
...
# specyfikacja nazwy pliku z komunikatami o błędach
#QCG stage-in-dir=.
...
# pliki danych będą brane z bieżącego katalogu
#QCG stage-out-dir=.
...
# pliki wynikowe będą umieszczane w bieżącym kagalogu
module load plgrid/apps/unres-e0ll2y/3.2.1
...
# ładowanie modułu zawierającego komp0onentu usługi dla pola E0LL2Y
export NPROCS=`cat $PBS_NODEFILE | wc -l`
cd $PBS_O_WORKDIR
mpirun -machinefile $PBS_NODEFILE -np $NPROCS unres.csh # uruchamiamie skryptu unres.csh
----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- Plik unres.csh
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------#!/bin/cshsetenv FGPROCS 1 # obliczenia energii nie są zrównoleglone
setenv POT
...
GB # specyfikacja potencjału oddziaływań między łańcuchami bocznymi (musi być taka dla pola E0LL2Y)
setenv PREFIX 1L2Y_lang # specyfikacja nazw plików; plik wynikowy będzie się nazywał 1L2Y_lang.inp
setenv OUT1FILE YES # plik wynikowy będzie produkowany tylko przez procesor główny (tutaj nie jest to ważne bo obliczenia są jednoprocesorowe)
unresMD_gfortran_MPICH_E0LL2Y.exe $* # uruchamianie aplikacji.
----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- Plik Plik danych 1L2Y_lang.inp (uwaga: formatowanie przy wyświetlaniu na stronie podręcznika nie jest poprawne); plik poprawnie sformatowany można pobrać stąd. Poniżej podany jest tylko przykładowy skrótowy opis; pełen opis danych znajduje się w dokumentacji pakietu UNRES (w języku angielskim).
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------...
1L2Y # tytuł (łańcuch tekstowy 80-znakowy).
SEED=-3059743 PDBREF
...
MD # ogólne dane; startowa liczba losowa, specyfikacja, że będzie struktura odniesienia i specyfikacja obliczeń MD
nstep=1000000 ntwe=100 ntwx=10000 dt=0.01 damax=10000.0 lang=1 scal_fric=0.01 &
...
# 10 mln kroków dynamiki, zrzut energii co 100 kroków, zrzut
...
współrzędnych co 10000 kroków, krok 0.01 jednostek "gruboziarnistych" (4.89 fs), obliczenia w trybie Langevina, skalowanie lepkości wody przez 1/100, kontynuacja tych danych w następnej linii ("&" w 80-tej kolumnie).
t_bath=...
300 # temperatura 300 K.
WLONG=1.00000 WSCP=1.23315 WELEC=0.84476 WBOND=1.00000 WANG=0.62954 &
...
# wagi wkładów do energii (nie mogą być zmienione dla używanej wersji pola siłowego)
WSCLOC=0.10554 WTOR=1.84316 WTORD=1.26571 WCORRH=0.19212 WCORR5=0.00000 &
WCORR6=0.00000 WEL_LOC=0.37357 WTURN3=1.40323 WTURN4=0.64673 WTURN6=0.00000 &
WVDWPP=0.23173 WHPB=1.00000 WSCCOR=0.0 &
CUTOFF=7.00000 WCORR4=0.00000
1L2Y.pdb
...
# nazwa pliku ze strukturą eksperymentalną (strukturą odniesienia); podlinkowany do pobrania.
...
22 # 22 reszty aminokwasowe
D ASN LEU TYR ILE GLN TRP LEU LYS ASP GLY GLY PRO SER SER GLY ARG PRO PRO PRO # sekwencja białka w kodzie trójliterowym (D oznacza grupę blokującą, która nie zawiera pełnej grupy peptydowej); format 20 (1X,A3).
SER D
...
0 # brak reszt cysteiny mogących tworzyć wiązania disulfidowe
...
0 # brak więzów
104.9286 87.6954 81.9040 82.4664 84.4658 83.9446 85.3958 91.
...
4490 # specyfikacja geometrii wirtualnej: wirtualne kąty walencyjne, wirtualne kąty torsyjne oraz kąty sferyczne określające geometrię łańcuchów bocznych.
94.6016 101.8618 119.3634 94.3628 96.2641 138.1186 96.2995 129.7022
109.4446 106.3486 106.0416 108.7182
-100.4302 72.4264 66.0265 51.4337 53.5083 60.5686 44.1766 67.6037
-72.8136 -61.9155 -75.8939 67.3579 129.7011 -95.5708 63.9717 -74.5043
-122.3059 -134.6048 92.1133
102.3561 120.0919 152.3636 134.9756 122.3426 152.1780 159.0518 100.5580
139.9612 0.0000 0.0000 117.4525 137.0250 146.2899 0.0000 93.9014
101.0251 113.0432 93.7775 153.8351
-82.3166 -56.6854 85.0901 -88.6662 -140.9453 38.0240 179.4707 -73.0903
-144.7967 0.0000 0.0000 -133.1635 -106.6593 -130.3055 0.0000 -102.7467
-111.6412 -122.0445 -102.3738 -143.3033
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Uruchamianie
. Po zalogowaniu się na serwer, z którego można uruchamiać zadania w systemie Qcg-Qoscos, np. qcg.man.poznan.pl i przeniesieniu tam wymienionych powyżej plików, należy wydać polecenie:
qcg-sub langdyn.qcg
Do monitorowania zadań służą polecenia qcg.
...
Polecenie konwersji dla podanego przykładu jest następujące:
xdrf2pdb three seq-3let 1L2Y_lang_MD000.cx 1 1 100 1L2Y_lang.pdb
gdzie
three oznacza, że podawana będzie sekwencja aminokwasowa białka w kodzie trójliterowym
seq-3let jest plikiem z sekwencją; format 20(A3, 1X); zawartość poniżej
D ASN LEU TYR ILE GLN TRP LEU LYS ASP GLY GLY PRO SER SER GLY ARG PRO PRO PRO
SER D
1L2Y_lang_MD000.cx jest nazwą pliku ze współrzędnymi konformacji białka z obliczonej trajektorii w formacie skompresowanym.
...
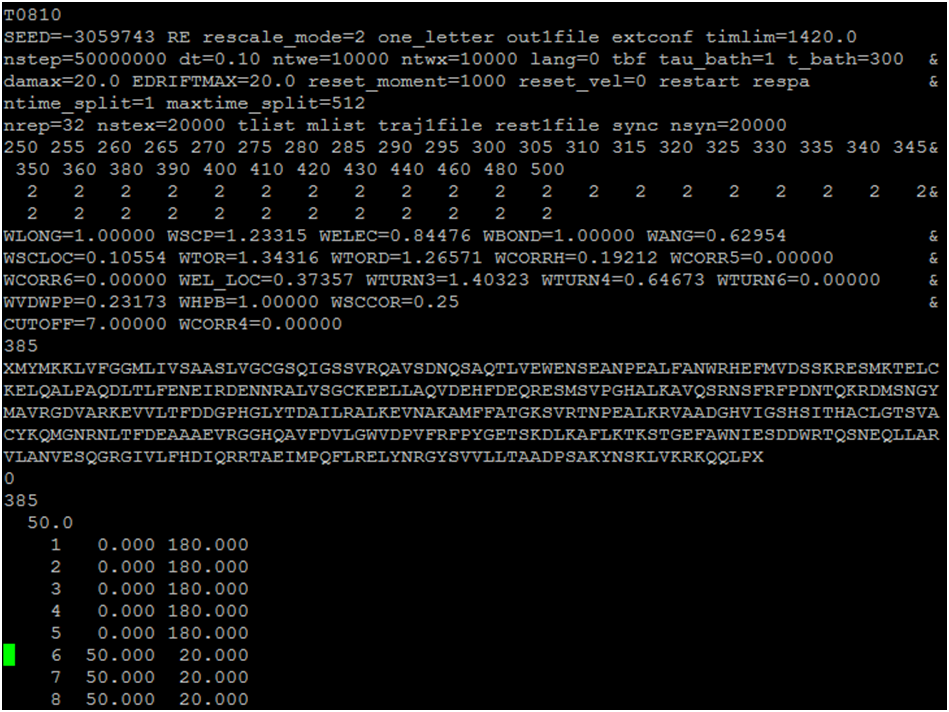
Pole siłowe UNRES umożliwia przeprowadzanie przewidywania de novo struktury białka, korzystając jedynie z sekwencji aminokwasowej. W tym celu należy wykonać symulację dynamiki molekularnej ze zwielokrotnioną wymianą replik (MREMD) wprowadzając sekwencję białka o poszukiwanej strukturze do przykładowego (Rysunek 1) pliku wejściowego MREMD.
Rysunek 1. Przykładowy plik wejściowy używamy w polu siłowym UNRES. Linia pierwsza stanowi komentarz, linie 2-14 zawierają parametry używane w polu siłowym, linia 15 zawiera całkowitą liczbę reszt aminokwasowych symulowanego układu z uwzględnieniem „dummy atom”, natomiast kolejne linie zawierają sekwencję aminokwasową w kodzie jednoliterowym wraz z „dummy atom” (litery X). Ostatnie linie wykorzystywane są do wprowadzenia ograniczeń swobody układu (więzów) np. na strukturę drugorzędową lub odległości między parami atomów.
Przy wprowadzaniu sekwencji należy pamiętać o dodawaniu „dummy atom” „dummy atom” na początku i końcu każdego łańcucha białka (litery X w kodzie jednoliterowym).
...
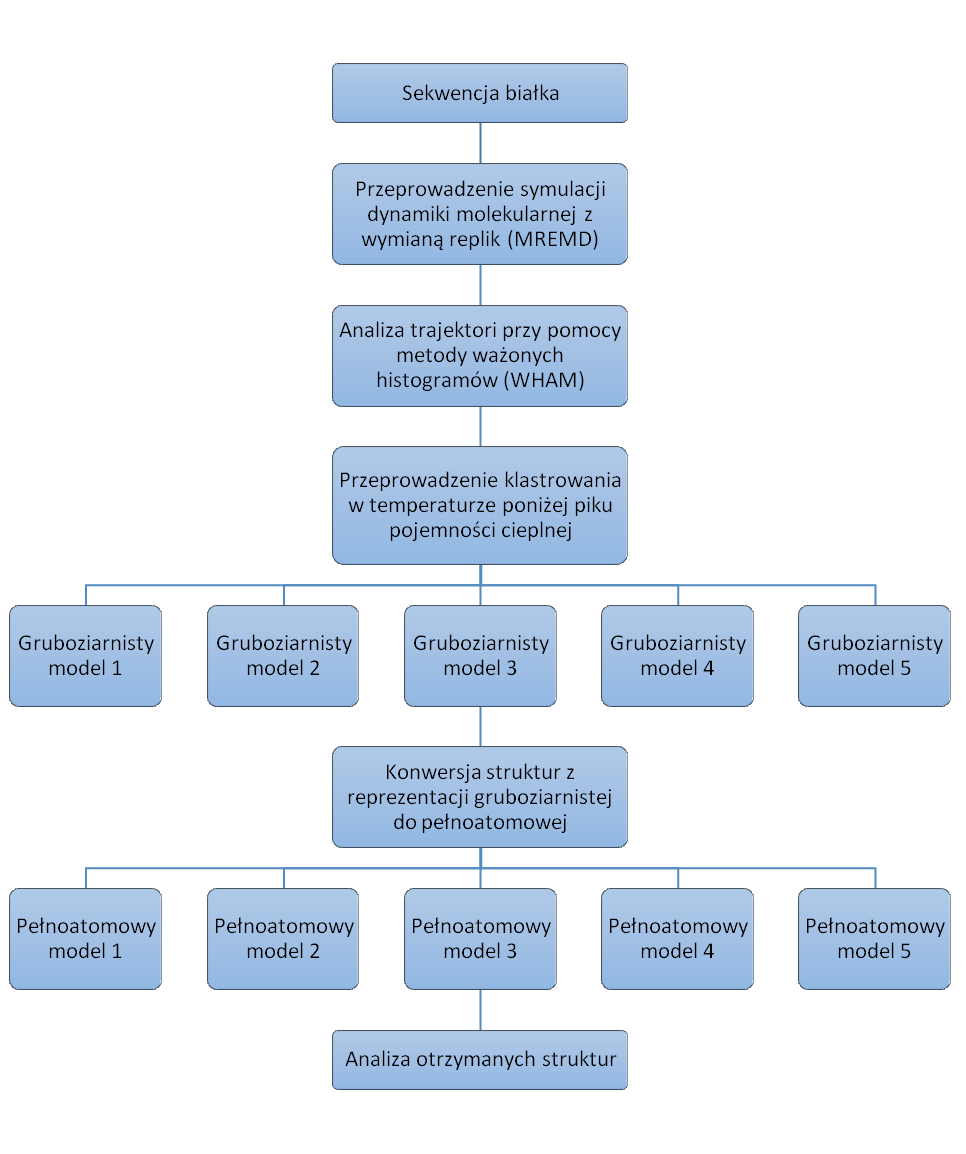
Zalecany schemat postępowania został umieszczony na poniższym diagramie:
Gdzie szukać dalszych informacji?
Strona dokumentacji pakietu UNRES ze szczegółowym opisem wprowadzanych danych (w języku angielskim).
| Info |
|---|
| Można też dodać sekcję "Co dalej?" ze wskazaniem (odnośnikiem) do dalszej części dokumentacji, o ile jest wymagana. |